Les projets
L’interféron, qui est produit par les cellules infectées suite à la détection d’agents pathogènes (virus ou bactéries), est la première ligne de défense contre les infections. L’interféron régule l’expression de plusieurs centaines de gènes, les gènes dits stimulés par l’interféron (ou ISG), à la fois dans les cellules infectées et dans les cellules avoisinantes. Cette reprogrammation transcriptionnelle induit la mise en place d’un état dit antiviral. La plupart des virus sont sensibles à cet état antiviral et incapables de se répliquer efficacement lorsque leurs cellules cibles ont prélablement été exposées à de l’interféron. C’est le cas notamment du virus de l’immunodéfience humaine de type 1 (VIH-1), du virus influenza A et du coronavirus à l’origine de la pandémie de COVID19, le SARS-CoV-2.
Les GTPases de haut poids moléculaires MX1 et MX2 joue un rôle significatif dans l’inhibition de la réplication virale induite par l’interféron. La protéine MX1 humaine présente une activité antivirale large et est capable d’inhiber de nombreux virus à ARN (dont le virus influenza A) et à ADN, à différentes étapes de leurs cycles réplicatifs. L’activité de son homologue MX2 semble plus étroite et restreinte notamment à certains lentivirus, dont le VIH-1. MX2 inhibe l’import nucléaire et l’intégration de l’ADN viral. MX1 et MX2 semblent reconnaître et intéragir avec des composants clés des complexes nucléoprotéiques viraux et inhiber ainsi la réplication virale. Cependant, le détail des mécanismes moléculaires mis en jeu restent à être élucidés. D’autres ISG inhibant le VIH-1 et le virus influenza A ont été identifiés, cependant nos données préliminaires montrent que des ISG additionnels restent à identifier. Nous avons notamment récemment identifié la courte isoforme de NCOA7 (NCOA7-AS) comme une protéine capable d’inhiber l’entrée de virus entrant par endocytose, dont le virus influenza A.
Notre équipe, initialement créée en Janvier 2015 grâce au soutien du programme ATIP-Avenir, s’intéresse à l’identification des effecteurs cellulaires de l’état antiviral et à la compréhension des mécanismes de restriction antivirale.
Nos différents projets sont les suivants :
- Compréhension du mécanisme d’action des protéines antivirales MX, NCOA7-AS et DDX42
- Identification et caractérisation de nouveaux effecteurs antiviraux induits par l’interféron
- Identification des gènes régulant la réplication des virus influenza et SARS-CoV-2 et recherche de nouveaux composés antiviraux
Nous employons des technologies de pointe, tels que des cribles CRISPR/Cas9 à l’échelle du génome, la protéomique, ou encore la microscopie Airyscan, pour comprendre le mode d’action des ISG antiviraux et en identifier de nouveaux. Depuis Mars 2020, nous étudions le SARS-COV-2 : nous caractérisons l’effet inhibiteur des interférons, identifions les facteurs cellulaires régulant sa réplication à l’aide de cribles CRISPR à l’échelle du génome et criblons des molécules et extraits naturels pour identifier de nouvelles pistes thérapeutiques.
Mots clés
Interféron, restriction antivirale, VIH-1, virus influenza A, SARS-CoV-2, immunité innée, signalisation, cribles génétiques, CRISPR/Cas9
Membres de l’équipe
- Caroline Goujon – CR1 INSERM

Après une formation technique (BTS et année de spécialisation en virologie moléculaire, ESTBA, Paris), Caroline a rejoint le magistère de biologie moléculaire et cellulaire (licence, maîtrise, DEA) de l’Ecole Normale Supérieure (ENS) de Lyon. Elle a effectué son DEA et sa thèse de doctorat dans le laboratoire du Dr Jean-Luc Darlix (Inserm U412/U758; Sept. 2002-Jan. 2008), sous la direction du Dr Andrea Cimarelli. Elle a étudié la restriction des cellules dendritiques humaines à l’infection par le VIH-1 et découvert que la protéine Vpx du VIH-2/SIV est capable de lever ce blocage. Elle a ensuite effectué un long post-doctorat dans le laboratoire du Prof. Michael Malim au King’s College London, au Royaume-Uni (Fev. 2008-Dec. 2014), où elle a obtenu un Marie Curie Intra-European Fellowship pour étudier la réponse des cellules hôtes à l’infection par le VIH-1. Elle s’est intéressée aux relations entre le VIH-1 et le système interféron et a caractérisé en détail la restriction du VIH-1 induite par l’interféron. Elle a notamment identifié la protéine MX2 humaine comme l’un des majeurs ISG inhibant le VIH-1. En 2014, elle a obtenu un poste CR1 INSERM et un financement ATIP-Avenir pour créer son groupe de recherche en France. Elle a intégré l’IRIM (ancien CPBS) en Janvier 2015. Caroline a depuis obtenu en 2016 des financements Sidaction et ANRS, en 2017 un financement ERC StG pour son projet ANTIViR et en 2019 un financement ERC PoC pour le projet FluAttack, mené par Olivier Moncorgé dans l’équipe. En 2020, l’équipe a obtenu plusieurs financements pour mener des recherches sur le SARS-CoV-2 de la part du programme MUSE de l’Université de Montpellier, de l’INSB et de la Fondation CNRS, ces deux derniers financements concernant des projets de cribles de molécules, portés par Olivier Moncorgé. En septembre 2020, l’équipe a obtenu deux financements ANR : un financement ANR-RACOVID pour l’identification de nouvelles cibles thérapeutiques contre le SARS-CoV-2 grâce à l’emploi de cribles CRISPR à l’échelle du génome, et un financement collaboratif pour comprendre le mode d’action de NCOA7-AS au niveau cellulaire et son rôle in vivo (collaboration avec M. Blaise, IRIM, et M. Wencker, CIRI). Depuis, Caroline a obtenu le soutien de l’I-Site de l’université de Montpellier avec un financement ERC tremplin puis l’ERC CoG 2022 pour le projet InVIRium, qui vise à utiliser des modèles pertinents d’épithéliums respiratoires primaires cultivés à l’interface air-liquide pour caractériser la réplication des virus respiratoires. En 2023, Caroline a également obtenu un financement collaboratif Horizon Europe dans le cadre du projet APPEAL (2024-2028) qui vise à développer de nouveaux antiviraux, et a été promue DR2 par l’Inserm.
- Olivier Moncorgé – IR CNRS (depuis Dec. 2021), ancien post-doctorant (ATIP – ANRS – ERC). Fev. 2015-Dec. 2020

Après avoir été responsable du groupe de biologie moléculaire dans une start-up française (Aptanomics SA, Lyon) puis ingénieur d’études (IE) sur la traduction du VIH à Ecole Normale Supérieure de Lyon (équipe du Dr T. Ohlmann), Olivier a effectué sa thèse de doctorat puis un post-doctorat de 2 ans dans le laboratoire du Prof. Wendy Barclay à Imperial College London (Royaume-Uni). Il a travaillé sur les bases moléculaires de la restriction des polymérases des virus influenza aviaires chez les mammifères. Pendant son premier post-doc, il a fait partie du consortium européen ANTIFLU, visant à identifier de nouveaux inhibiteurs de la réplication du virus influenza A. Olivier a rejoint l’équipe en février 2015, à l’aide du support de l’ANRS (financement de 2 ans obtenu par C. Goujon en liaison avec l’ATIP-Avenir, puis renouvellement d’1 an obtenu par O. Moncorgé). Olivier a travaillé à partir de mars 2018 sur le projet ERC ANTIViR et d’avril 2019 sur le projet ERC PoC FluAttack et est porteur des projets relatifs au virus influenza A. Grâce à son expertise sur les virus respiratoires, l’équipe a rapidement pu se mettre à travailler sur le SARS-CoV-2 en Mars 2020. Olivier a notamment mené tous les projets de criblage de molécules contre le SARS-CoV-2. Le contrat d’Olivier à l’IRIM s’est terminé en Dec. 2020. En 2021 Olivier s’est établi en tant qu’auto-entrepreneur et a fait du consulting sur les virus respiratoires. En 2021, Olivier a obtenu un poste d’IR spécialiste des virus respiratoires au CNRS et est revenu à l’IRIM et dans l’équipe au 1er décembre 2021. Au sein de l’IRIM et en parallèle de son travail dans l’équipe, Olivier a développé un plateau technique d’étude des virus respiratoires (page web en construction). Le but de ce plateau est de partager l’expertise d’Olivier sur les virus respiratoires et en particuler les virus influenza A, B et les coronavirus, de former les personnes de l’institut à leur manipulation en BSL-2 et BSL-3, de développer de nouveaux systèmes rapporteurs viraux innovants et de les partager avec la communauté scientifique sous forme de collaboration ou prestation de service, notamment pour du criblage de molécules antivirales.
- Mary Arnould – PRAG (université de Montpellier) avec une convention d’accueil en tant que chercheuse

Normalienne aggrégée (ENS-Cachan), Mary a effectué sa thèse à l’université Paris XI en immunologie et enseigne depuis à l’IUT de Montpellier. Mary a rejoint l’équipe en mars 2019 pour apporter son expertise de biochimiste et travailler sur la caractérisation de l’activité antivirale de MX1, de NCOA7-AS et de DDX42, en étroite collaboration avec Mickaël Blaise (IRIM).
- Antoine Rebendenne – Post-doctorant

Normalien agrégé en Biochimie-Génie Biologique (BGB) de l’Ecole Normale Supérieure Paris-Saclay (ex ENS Cachan), Antoine a réalisé le Master 2 de Virologie Fondamentale (Sorbonne Université/Institut Pasteur). Après avoir effectué différents stages à la fois en France (au sein même de l’équipe durant l’été 2016) mais aussi à Londres (Imperial College London, UK) en 2017 et à New York (Rockefeller University, USA) en 2018-2019, il est revenu dans l’équipe pour son stage de M2R de décembre 2019 à fin juillet 2020. Sous la supervision de Caroline, Antoine a travaillé sur la caractérisation d’une nouvelle protéine antivirale induite par l’interféron, NCOA7-AS, et entre Mai 2020 et Janvier 2022, a développé plusieurs projets sur le SARS-CoV-2. Antoine a effectué sa thèse à la fois dans l’équipe et en cotutelle avec Mélanie Wencker (CIRI) avec laquelle il a étudié le rôle de NCOA7-AS in vivo. Antoine a soutenu sa thèse en Novembre 2023 et est revenu dans l’équipe en tant que post-doctorant en Février 2024 afin de mener le projet ERC InVIRium.
- Elodie Bishop. Doctorante
Elodie est étudiante en double cursus Médecine-Sciences de l’ENS de Paris. Elle effectue une césure dans ses études de Médecine pour s’investir dans la recherche en virologie. Elle a réalisé le Master 2 de Virologie Fondamentale de Sorbonne Université et l’Institut Pasteur. Grâce à l’obtention d’un CDSN, elle est en thèse dans l’équipe depuis Septembre 2022. Elle travaille sur la caractérisation d’une nouvelle protéine inhibitrice des coronavirus SARS-CoV-2 et HCoV-229E.
- Alice Trausch. Doctorante CIFRE

Alice effectue une thèse CIFRE entre le laboratoire et l’entreprise Voxcan à Lyon. Le but de sa thèse est de développer de nouveaux systèmes de génétiques inverses pour les virus respiratoires (Influenza et SARS-CoV-2), sous la direction d’Olivier Moncorgé, et de mettre en place des systèmes de cultures primaires d’épithéliums respiratoires.
- Jimmy Cadènes – Doctorant

Jimmy est diplômé de l’école d’ingénieur AgroParisTech, et a réalisé le Master 2 de Virologie Fondamentale de l’Institut Pasteur/Université Paris-Cité. Il a effectué un stage de 4 mois à l’Institut Pasteur en 2022 sur la mise au point de systèmes de génétique inverse et de pseudotypage d’un vésiculovirus de chauve-souris, et un stage de 6 mois en 2023 à l’ENS de Lyon sur l’impact du virus herpes simplex 1 sur l’interactome des ribosomes. Il a aussi effectué en 2022-2023 un stage de virologie « de terrain » de 4 mois à l’Institut Pasteur du Cambodge, sur un coronavirus aviaire (l’infectious bronchitis virus). Il a ensuite rejoint l’équipe en 2024 pour son stage de Master 2 pendant lequel il a travaillé sur le mécanisme d’action de la protéine antivirale à large spectre MX1 contre différents virus humains : le virus de La Crosse (LaCV), le virus de la stomatite vésiculeuse (VSV) et le virus influenza A (IAV). Grâce à l’obtention d’un financement FRM (Fondation pour la Recherche Médicale), Jimmy réalise désormais sa thèse sur l’étude de la restriction du virus influenza A au sein des cellules épithéliales respiratoires humaines, avec un focus sur la protéine MX1.
- Issy Fernanda Hincapie Mena – Ingénieure d’études

Fernanda a rejoint l’équipe de Caroline Goujon pour la première fois en janvier 2024, dans le cadre de son stage de Master 2 en Biologie, parcours Microbiology, Immunobiology and Infectious Diseases (MIDI) à l’Université Grenoble Alpes (UGA). Lors de ce stage, elle a travaille avec Olivier Moncorgé et Alice Trausch sur le développement de virus influenza rapporteurs pour des souches ayant circulé récemment, en utilisant la technique de génétique inverse.En septembre 2024, Fernanda a intégré l’équipe en tant qu’ingénieure d’études dans le cadre du projet APPEAL (Antivirus Pandemic Preparedness European pLatform). Elle contribue à l’optimisation de techniques d’édition génomique basées sur le système CRISPR, afin de valider des gènes d’intérêt impliqués dans la réplication de virus pathogènes majeurs et qui permettraient de mieux comprendre les interactions entre virus et hôte. Ces travaux s’inscrivent dans la stratégie globale du projet, visant à identifier des facteurs clés chez l’hôte et à développer des antiviraux à large spectre contre des virus pathogènes émergents ou ré-émergents.
- Guorong Sun – Post-Doctorant
Guorong a obtenu son doctorat à l’Institut de virologie de l’École de médecine de Hanovre, en Allemagne, dans le laboratoire du professeur Abel Viejo-Borbolla, et a ensuite effectué un court stage postdoctoral dans le même laboratoire. Au cours de cette période, les recherches de Guorong se sont principalement concentrées sur les mécanismes de neurotropisme du virus de l’herpès simplex. Depuis juillet 2024, Guorong a rejoint l’équipe de du Dr Caroline Goujon en tant que postdoc pour travailler sur le projet InVIRium financé par l’ERC.
- Justine Lagisquet – Post-Doctorante
- Lenka Kocianova – Étudiante de M2

Lenka a rejoint l’équipe pour la première fois pour deux courts stages lors de sa Licence, en 2022 et 2023. Par la suite, ayant effectué deux stages, à l’Institut Cochin à Paris et à l’Institut de virologie de l’Université de Marburg, en Allemagne, lors de son Master en infectiologie à l’Université de Montpellier, elle a décidé de nous rejoindre à nouveau pour son stage de Master 2. Elle travaille actuellement avec Caroline, Mary, Jimmy et Olivier sur un projet s’intéressant à l’interface entre la signalisation cellulaire et la GTPase MX1 lors de l’infection par le virus influenza, dans le but de contribuer à une meilleure compréhension de rôle antiviral général de ce facteur de restriction.
L’équipe Interferon et Restriction Antivirale en mars 2025

L’équipe Interferon et Restriction Antivirale en mars 2023

De gauche à droite : I. Fernanda Hincapie Mena, Alice Trausch, Jimmy Cadènes, Elodie Bishop, Inken Junod, Ana-Luiza Chaves Valadao (ex IR), Marine Tauziet (ex IE), Olivier Moncorgé, Caroline Goujon, Mary Arnould, Fransisco Garcia de Gracia (ex post doc), Antoine Rebendenne.
L’équipe Interféron et Restriction Antivirale en septembre 2022
 De gauche à : Francisco Garcia de Gracia, Caroline Goujon, Elodie Bishop (bottom row), Marine Tauziet (top row), Joe McKeller, Olivier Moncorgé, Ana Luise Chaves Valadão, Mary Arnould.
De gauche à : Francisco Garcia de Gracia, Caroline Goujon, Elodie Bishop (bottom row), Marine Tauziet (top row), Joe McKeller, Olivier Moncorgé, Ana Luise Chaves Valadão, Mary Arnould.
L’équipe Interféron et Restriction Antivirale en été 2020.
 De gauche à droite: Francisco Garcia de Gracia, Olivier Moncorgé, Marine Tauziet, Joe McKellar, Caroline Goujon, Mary Arnould, Ana Luisa Chaves Valadão, Antoine Rebendenne, Boris Bonaventure.
De gauche à droite: Francisco Garcia de Gracia, Olivier Moncorgé, Marine Tauziet, Joe McKellar, Caroline Goujon, Mary Arnould, Ana Luisa Chaves Valadão, Antoine Rebendenne, Boris Bonaventure.
L’équipe Interféron et Restriction Antivirale en Décembre 2019.
 De gauche à droite : A. Rebendenne, M. Arnould, F. Garcia de Gracia, M. Tauziet, O. Moncorgé, B. Bonaventure, J. McKellar, and C. Goujon.
De gauche à droite : A. Rebendenne, M. Arnould, F. Garcia de Gracia, M. Tauziet, O. Moncorgé, B. Bonaventure, J. McKellar, and C. Goujon.
L’équipe en juillet 2019
 From left to right: M. Arnould, O. Moncorgé, J. McKellar, M. Tauziet, F. Garcia de Gracia, B. Bonaventure, C. Goujon, S. Allahverdiyeva.
From left to right: M. Arnould, O. Moncorgé, J. McKellar, M. Tauziet, F. Garcia de Gracia, B. Bonaventure, C. Goujon, S. Allahverdiyeva.
L’équipe au printemps 2019 
De gauche à droite : B. Bonaventure, J. McKellar, C. Goujon, C. André, S. Allahverdiyeva, O. Moncorgé, M. Tauziet, M. Arnould
L’équipe au printemps 2018 
De gauche à droite : O. Moncorgé, W. Djilli, M. Tauziet, B. Bonaventure, C. Goujon, C. André, J. McKellar
L’équipe en Octobre 2017

L’équipe aux Journées IRIM 2017. De gauche à droite : O. Moncorgé, B. Bonaventure, C. Goujon, M. Tauziet.
L’équipe en Juillet 2017

De gauche à droite : R. Planès, C. Goujon, M. Tauziet, B. Bonaventure, O. Moncorgé, E. Partiot.
L’équipe en Septembre 2016

L’équipe aux Journées IRIM 2016. De gauche à droite : B. Bonaventure, C. Goujon, O. Moncorgé, R. Planès.
Anciens membres et étudiants de l’équipe
- Inken Junod – Etudiante de M2
 Inken est étudiante en Master 2 « Molecular life sciences Master » à l’Université de Hambourg, Allemagne.Elle travaille avec Olivier Moncorgé et Alice Trausch sur l’optimisation de la detection des virus Influenza produits par génétique inverse.
Inken est étudiante en Master 2 « Molecular life sciences Master » à l’Université de Hambourg, Allemagne.Elle travaille avec Olivier Moncorgé et Alice Trausch sur l’optimisation de la detection des virus Influenza produits par génétique inverse.
- Ana Luiza Chaves Valadao – Ingénieure de Recherche (ERC PoC FluAttack puis ERC StG ANTIViR)
 Brésilienne, Ana a étudié les sciences biologiques à l’Université d’Etat de Rio de Janeiro. Elle a obtenu un Master de Parasitologie de la Fondation Oswaldo Cruz, au cours duquel elle a participé au développement d’un vaccin bivalent fièvre jaune/dengue. Pendant son doctorat, elle s’est focalisée sur le rôle des P-Bodies et granules de stress au cours de la réplication du VIH-1. Pendant sa thèse, elle a également passé plusieurs mois dans l’équipe de Monsef Benkirane à l’Institut de Génétique Humaine (Montpellier), où elle a étudié la régulation de l’activité du facteur de restriction SAMHD1. Elle a ensuite réalisé un postdoc au Brésil, sur la latence du VIH-1 et l’identification de composés antiviraux contre le virus Zika. Au cours de son postdoc, Ana a également enseigné en licence à l’Université Fédérale de Rio de Janeiro. En 2016, elle revient à l’IGH et rejoint l’équipe Nadine Laguette en tant que postdoc, pour travailler sur l’inflammation liée au cancer (MSDAvenir fellowship). Après deux années de postdoc, Ana devient Ingénieure de recherche dans la même équipe. Ana a rejoint l’équipe en plein confinement, en Avril 2020, tout d’abord pour prêter main forte sur les projets SARS-CoV-2 et l’identification de nouveaux antiviraux. Le premier projet d’Ana a visé à caractériser de nouveaux composés antiviraux contre influenza (projet ERC PoC FluAttack), sous la direction d’Olivier Moncorgé. Depuis, Ana participe aux nombreux autres projets de l’équipe, sur le SARS-CoV-2 notamment et elle mène également un projet sur l’étude des gènes humains régulant la réplication d’IAV (ERC StG ANTIViR). Depuis 2023, elle est passée Ingénieure à l’université pour travailler sur le projet InVIRium.
Brésilienne, Ana a étudié les sciences biologiques à l’Université d’Etat de Rio de Janeiro. Elle a obtenu un Master de Parasitologie de la Fondation Oswaldo Cruz, au cours duquel elle a participé au développement d’un vaccin bivalent fièvre jaune/dengue. Pendant son doctorat, elle s’est focalisée sur le rôle des P-Bodies et granules de stress au cours de la réplication du VIH-1. Pendant sa thèse, elle a également passé plusieurs mois dans l’équipe de Monsef Benkirane à l’Institut de Génétique Humaine (Montpellier), où elle a étudié la régulation de l’activité du facteur de restriction SAMHD1. Elle a ensuite réalisé un postdoc au Brésil, sur la latence du VIH-1 et l’identification de composés antiviraux contre le virus Zika. Au cours de son postdoc, Ana a également enseigné en licence à l’Université Fédérale de Rio de Janeiro. En 2016, elle revient à l’IGH et rejoint l’équipe Nadine Laguette en tant que postdoc, pour travailler sur l’inflammation liée au cancer (MSDAvenir fellowship). Après deux années de postdoc, Ana devient Ingénieure de recherche dans la même équipe. Ana a rejoint l’équipe en plein confinement, en Avril 2020, tout d’abord pour prêter main forte sur les projets SARS-CoV-2 et l’identification de nouveaux antiviraux. Le premier projet d’Ana a visé à caractériser de nouveaux composés antiviraux contre influenza (projet ERC PoC FluAttack), sous la direction d’Olivier Moncorgé. Depuis, Ana participe aux nombreux autres projets de l’équipe, sur le SARS-CoV-2 notamment et elle mène également un projet sur l’étude des gènes humains régulant la réplication d’IAV (ERC StG ANTIViR). Depuis 2023, elle est passée Ingénieure à l’université pour travailler sur le projet InVIRium.
- Francisco Garcia de Gracia – Postdoc (ERC)
 De nationalité chilienne, Francisco a un diplôme d’ingénieur en biotechnologie moléculaire de l’université du Chili. Son travail a alors été centré sur la caractérisation du complexe de pre-intégration du virus de la leucémie murine dans le laboratoire du Prof. Monica Acevedo. Francisco a ensuite réalisé sa thèse de doctorat en microbiologie à l’Université de Chile-Université de Santiago et a travaillé dans le laboratoire du Prof. Ricardo Soto-Rifo. Sa thèse s’est focalisé sur la régulation de l’expression des gènes du VIH. Francisco a travaillé sur le projet ANTIViR de Mai 2019 à Janvier 2023.
De nationalité chilienne, Francisco a un diplôme d’ingénieur en biotechnologie moléculaire de l’université du Chili. Son travail a alors été centré sur la caractérisation du complexe de pre-intégration du virus de la leucémie murine dans le laboratoire du Prof. Monica Acevedo. Francisco a ensuite réalisé sa thèse de doctorat en microbiologie à l’Université de Chile-Université de Santiago et a travaillé dans le laboratoire du Prof. Ricardo Soto-Rifo. Sa thèse s’est focalisé sur la régulation de l’expression des gènes du VIH. Francisco a travaillé sur le projet ANTIViR de Mai 2019 à Janvier 2023.
- Joe McKellar – Postdoc (ancien doctorant, université de Montpellier)
 De nationalité britannique, Joe a rejoint le Master de Microbiologie et Immunologie de l’université de Montpellier après une licence de biologie cellulaire et physiologie à l’université de Limoges. Il a effectué un stage de Master 2 dans l’équipe de Février à Juillet 2018, a été recruté en tant qu’IE en septembre et effectue depuis Octobre 2018 sa thèse de doctorat dans l’équipe depuis grâce à l’obtention de la bourse doctorale CBS2. Sous la supervision d’Olivier, Joe étudie le mécanisme d’action de la protéine MX1 contre le virus influenza A. Il s’est spécialisé en biologie cellulaire et microscopie. Joe a obtenu un financement de la FRM pour une 4e année de thèse. Joe a soutenu sa thèse le 17 novembre 2022 et après être resté quelques mois supplémentaires dans l’équipe, il a rejoint l’équipe de Karim Majzoub pour son post-doctorat.
De nationalité britannique, Joe a rejoint le Master de Microbiologie et Immunologie de l’université de Montpellier après une licence de biologie cellulaire et physiologie à l’université de Limoges. Il a effectué un stage de Master 2 dans l’équipe de Février à Juillet 2018, a été recruté en tant qu’IE en septembre et effectue depuis Octobre 2018 sa thèse de doctorat dans l’équipe depuis grâce à l’obtention de la bourse doctorale CBS2. Sous la supervision d’Olivier, Joe étudie le mécanisme d’action de la protéine MX1 contre le virus influenza A. Il s’est spécialisé en biologie cellulaire et microscopie. Joe a obtenu un financement de la FRM pour une 4e année de thèse. Joe a soutenu sa thèse le 17 novembre 2022 et après être resté quelques mois supplémentaires dans l’équipe, il a rejoint l’équipe de Karim Majzoub pour son post-doctorat.
- Boris Bonaventure – M2, doctorant puis postdoc (Fév. 2016-Sept. 2021)
 Boris a étudié à l’ENS de Lyon. Après des stages à l’université d’Oxford et à l’Harvard Medical School, Boris a rejoint l’équipe pour son troisième stage de Master en Février 2016. Il a ensuite travaillé en tant qu’ingénieur d’études dans l’équipe pendant l’été 2016. Classé 2e au concours de l’école doctorale CBS2, il a obtenu la bourse du gouvernement français MNERT pour réaliser sa thèse de doctorat dans l’équipe. Boris a commencé sa thèse en octobre 2016. Il a employé des cribles génétiques CRISPR/Cas9 à l’échelle du génome humain pour identifier de nouveaux inhibiteurs du VIH-1 régulés par l’interféron et a récemment identifié le gène humain DDX42 comme un inhibiteur intrinsèque du VIH-1 et des rétrotransposons ainsi que d’autres virus. Boris a obtenu une 4e année de financement de thèse auprès de la FRM et a soutenu sa thèse en Déc. 2020. Boris a ensuite enchainé avec un court postdoc (9 mois) dans l’équipe, financé par la Région Occitanie. Il est maintenant en postdoc aux USA, à Mount Sinai.
Boris a étudié à l’ENS de Lyon. Après des stages à l’université d’Oxford et à l’Harvard Medical School, Boris a rejoint l’équipe pour son troisième stage de Master en Février 2016. Il a ensuite travaillé en tant qu’ingénieur d’études dans l’équipe pendant l’été 2016. Classé 2e au concours de l’école doctorale CBS2, il a obtenu la bourse du gouvernement français MNERT pour réaliser sa thèse de doctorat dans l’équipe. Boris a commencé sa thèse en octobre 2016. Il a employé des cribles génétiques CRISPR/Cas9 à l’échelle du génome humain pour identifier de nouveaux inhibiteurs du VIH-1 régulés par l’interféron et a récemment identifié le gène humain DDX42 comme un inhibiteur intrinsèque du VIH-1 et des rétrotransposons ainsi que d’autres virus. Boris a obtenu une 4e année de financement de thèse auprès de la FRM et a soutenu sa thèse en Déc. 2020. Boris a ensuite enchainé avec un court postdoc (9 mois) dans l’équipe, financé par la Région Occitanie. Il est maintenant en postdoc aux USA, à Mount Sinai.
- Marine Tauziet – Ingénieure d’Etude, IE, CNRS (ex AI, ERC)
 Marine a travaillé en tant qu’AI puis IE dans l’équipe de 2017 à 2022. Elle a soutenu le diplôme de l’EPHE en Nov. 2021 et est maintenant en CDD à l’IRCM.
Marine a travaillé en tant qu’AI puis IE dans l’équipe de 2017 à 2022. Elle a soutenu le diplôme de l’EPHE en Nov. 2021 et est maintenant en CDD à l’IRCM.
- Oriane Pourcelot, étudiante en M2, université de Montpellier
- Laura Navar. Printemps 2020. Etudiante en L3 Pro, IUT Montpellier.
- Sona Allahverdiyeva. Printemps 2019. Etudiante en master (M2), Université de Montpellier. Sona effectue sa thèse de doctorat en virologie à Amsterdam.
- Charlotte Arpin-André, AI CNRS. Printemps 2018- Printemps 2019.
- Timothée Lalo-Deltour – Etudiant en L3. Eté 2018 (7 semaines).
- Joe McKellar – Etudiant en M2, Université de Montpellier (6 mois). Joe est resté en thèse.
- Wassila Djilli – Etudiante en M2, université de Strasbourg (5 mois).
 Après un BTS bio-analyses, Wassila a continué ses études en licence puis en Master biologie des microorganismes à l’Université de Strasbourg. Elle effectue un stage de Master 2 dans l’équipe de Janvier à Juin 2018. Wassila a travaillé avec Boris sur l’identification et la caractérisation de nouveaux inhibiteurs du VIH-1 induits par l’interféron. Après avoir travaillé en tant qu’ingénieure, Wassila a repris des études pour devenir pharmacienne.
Après un BTS bio-analyses, Wassila a continué ses études en licence puis en Master biologie des microorganismes à l’Université de Strasbourg. Elle effectue un stage de Master 2 dans l’équipe de Janvier à Juin 2018. Wassila a travaillé avec Boris sur l’identification et la caractérisation de nouveaux inhibiteurs du VIH-1 induits par l’interféron. Après avoir travaillé en tant qu’ingénieure, Wassila a repris des études pour devenir pharmacienne.
- Rémi Planès – Post-doctorant (Sidaction). Oct. 2015-Sept. 2017
Rémi a effectué sa thèse de doctorat à Toulouse (INSERM U1043, CHU Purpan), sous la direction du Prof. E. Bahraoui. Sa thèse a mis en évidence le rôle de la protéine Tat du VIH-1 dans la signalisation du TLR4. Rémi a ensuite obtenu un fellowship FRM de 1 an pour joindre l’équipe du Dr P. Guermonprez au Centre for Molecular and Cellular Biology of Inflammation (CMCBI), au King’s College London, où il a étudié la réponse des cellules dendritiques humaines aux hématies infectées par Plasmodium falciparum. Il a obtenu un financement post-doctoral de 2 ans auprès de Sidaction pour rejoindre l’équipe Interféron et restriction antivirale et étudier le rôle potentiel de MX2 dans la signalisation de l’immunité innée d’octobre 2015 à septembre 2017. Après un post-doctorat dans l’équipe d’Etienne Meunier à l’IPBS, Rémi est maintenant en CDI chez InvivoGen à Toulouse.
- Emma Partiot, étudiante en Master (M1). Université Paris Diderot University. Juin-Juillet 2017 (2 mois). Emma est en thèse dans l’équipe de Raphaël Gaudin, à l’IRIM.
- Leila Makrini, AI CNRS. Décembre-Juin 2017 (6 mois).
- Marion Chateauraynaud, étudiante en Master (M1). Université de Montpellier. Printemps 2017 (2 mois).
- Antoine Rebendenne, étudiant en L3. ENS-Cachan. Eté 2016 (2,5 mois). Antoine est revenu en M2 (M2 de virologie fondamentale, Université Paris & institut Pasteur, 8 mois) de Dec. 2019 à Juillet 2020 et effectue sa thèse dans l’équipe.
- Océane Paris, étudiante en Master (M1) student. Université de Montpellier. Printemps 2016 (2 mois).
- Boris Bonaventure, étudiant en Master (M2). Ecole Normale Supérieure de Lyon. Printemps 2016 (3 mois). Boris est resté en thèse dans l’équipe puis pour un court postdoc.
Main publications and recent preprints (C. Goujon)
# corresponding authors
* contributed equally to the experimental work
(bold: lab members)
2023
- McKellar J., Arnaud-Arnould M., Chaloin L., Tauziet M., Arpin-André C., Pourcelot O., Blaise M., Moncorgé O.#, and Goujon C.# An evolutionarily conserved N-terminal leucine is essential for MX1 GTPase antiviral activity against different families of RNA viruses. Journal of Biological Chemistry, in press, 2023.
2022
- McKellar J., Arnaud-Arnould M., Chaloin L., Tauziet M., Arpin-André C., Pourcelot O., Blaise M., Moncorgé O.#, and Goujon C.# An evolutionarily conserved N-terminal leucine is essential for MX1 GTPase antiviral activity against different families of RNA viruses. Journal of Biological Chemistry, in press, 2022.
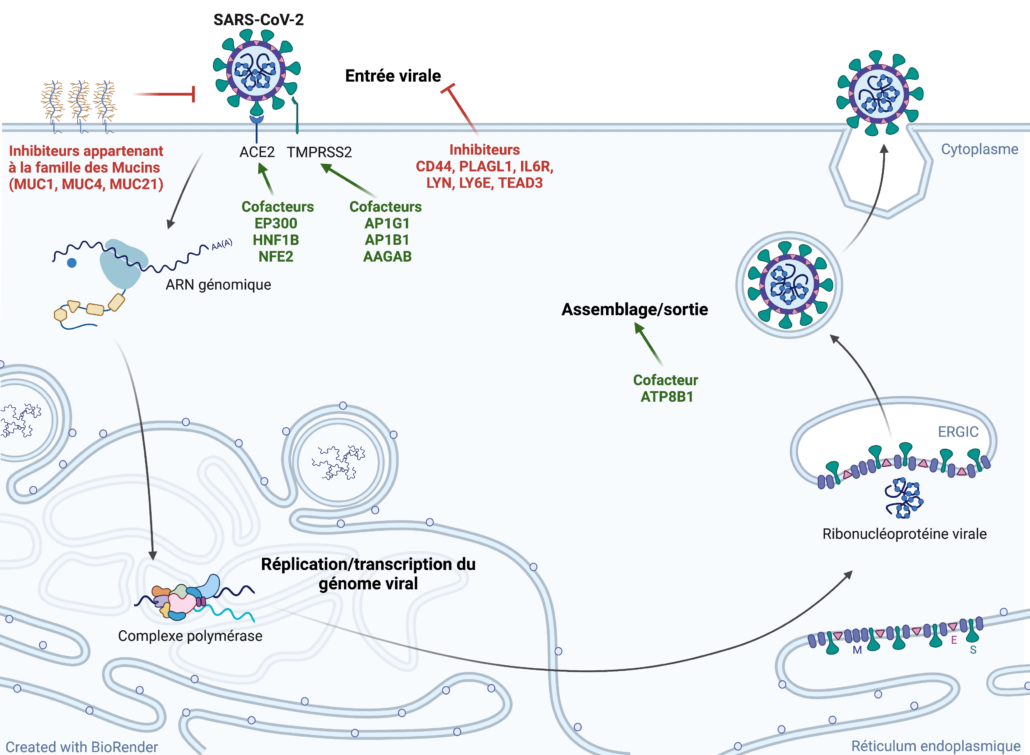
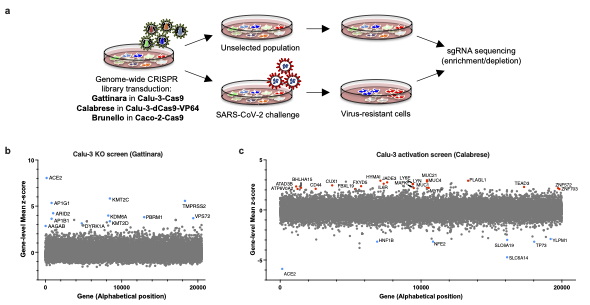
- Rebendenne A., Roy P., Bonaventure B.*, Chaves Valadão A.L.*, Desmarets L., Rouillé Y., Tauziet M., Arnaud-Arnould M., Giovannini D., Lee Y., DeWeirdt P., Hegde M., Garcia de Gracia F., McKellar J., Wencker M., Dubuisson J., Belouzard S., Moncorgé O., Doench J.G#, and Goujon C.# Bidirectional genome-wide CRISPR screens reveal host factors regulating SARS-CoV-2, MERS-CoV and seasonal HCoVs. Nature Genetics, 2022. 54(8):1090-1102
- Bonaventure B.#, and Goujon C.# DExH/D-box helicases at the frontline of intrinsic and innate immunity against viral infections. Journal of General Virology, 2022. 103(8). Review
2021
- Jouvenet N.#, Goujon C.#, Banerjee A.#. Clash of the titans: interferons and SARS-CoV-2. Trends in immunology, online Oct. 2021. doi: 10.1016/j.it.2021.10.009
- Arnaud-Arnould M., Tauziet M., Moncorgé O., Goujon C., Blaise M. Crystal structure of the TLDc domain of human NCOA7-AS. Acta Crystallographica Section F: Structural Biology Communications, 2021, 77(8).
- Bauby H., Ward C.C., Hugh-White R., Swanson C.M., Schulz R., Goujon C.# and Malim M.H.# HIV-1 Vpr Induces Widespread Transcriptomic Changes in CD4 + T Cells Early Postinfection. mBio, 2021 29;12(3):e0136921.
- Maarifi G., Lagisquet J., Hertel Q., Bonaventure B., Chamontin C., Fuchs K., Moncorgé O., Tauziet M., Mombled M., Papin L., Molès J.P. Bodet C., Lévèque N., Gross A., Arhel N., Nisole S., Van de Perre P., Goujon C., Blanchet F.P. Alarmin S100A9 restricts retroviral infection by limiting reverse transcription in human dendritic cells. EMBO Journal, 2021, 40(16):e106540.
- Rebendenne Antoine, Roy Priyanka, Bonaventure Boris*, Chaves Valadão Ana Luiza*, Desmarets Lowiese, Rouillé Yves, Tauziet Marine, Arnaud-Arnould Mary, Giovannini Donatella, Lee Yenarae, DeWeirdt Peter, Hegde Mudra, Garcia de Gracia Francisco, McKellar Joe, Wencker Mélanie, Dubuisson Jean, Belouzard Sandrine, Moncorgé Olivier, Doench John G.#, and Goujon Caroline#. Bidirectional genome-wide CRISPR screens reveal host factors regulating SARS-CoV-2, MERS-CoV and seasonal HCoVs. bioRxiv. https://www.biorxiv.org/content/10.1101/2021.05.19.444823v2
- Rebendenne A.*, Chaves Valadão A.L.*, Tauziet M.*, Maarifi G., Bonaventure B., Planès R., McKellar J., Nisole S., Arnaud-Arnould M., Moncorgé O.*#, and Goujon C.*# (2021). SARS-CoV-2 triggers an MDA-5 dependent interferon response which is unable to control replication in lung epithelial cells. Journal of Virology. https://jvi.asm.org/content/early/2021/01/28/JVI.02415-20
- Pila-Castellanos I., Molino D., McKellar J., Lines L., Da Graca J., Tauziet M., Chanteloup L., Mikaelian I., Meyniel-Schicklin L., Codogno P., Vonderscher J., Delevoye C., Moncorgé O., Meldrum E., Goujon C., Morel E., de Chassey B. Mitochondrial morphodynamics alteration induced by influenza virus infection as a new antiviral strategy. PloS Pathogens. 2021, DOI:10.1371/journal.ppat.1009340
2020
- Bonaventure B., Rebendenne A., Garcia de Gracia F. , Tauziet M., McKellar J., Chaves Valadão A.L., Courgnaud V., Bernard E., Briant L., Gros N., Djilli W., Arnaud-Arnould M., Parrinello H., Rialle S., Moncorgé O., and Goujon C.# (2020) A genome-wide CRISPR/Cas9 knock-out screen identifies the DEAD box RNA helicase DDX42 as a broad antiviral inhibitor. bioRxiv. doi: https://doi.org/10.1101/2020.10.28.359356
- Rebendenne A.*, Chaves Valadão A.L.*, Tauziet M.*, Maarifi G., Bonaventure B., Planès R., McKellar J., .Nisole S., Arnaud-Arnould M., Moncorgé O.*#, and Goujon C.*# (2020). SARS-CoV-2 replication triggers an MDA-5-dependent interferon production which is unable to efficiently control replication. bioRxiv. doi: https://doi.org/10.1101/2020.10.28.358945
2018
- Doyle T., Moncorgé O., Bonaventure B., Pollpeter D., Lussignol M., Tauziet M., Apolonia L., Catanese M.T., Goujon C.# and Malim M.H.# (2018). The interferon inducible isoform of NCOA7 inhibits endosome-mediated viral entry. Nature Microbiology, (12):1369-1376.
- Dicks MDJ, Betancor G, Jimenez-Guardeño JM, Pessel-Vivares L, Apolonia L, Goujon C, Malim MH. (2018) Multiple components of the nuclear pore complex interact with the amino-terminus of MX2 to facilitate HIV-1 restriction. PLoS Pathogens. 14(11):e1007408.
2016
- Dicks, M., Goujon, C., Pollpeter D., Betancor G., Apolonia L., Bergeron J.R., Malim M.H. (2016) Oligomerization requirements for MX2 mediated suppression of HIV-1 infection. Journal of Virology 90 (1), 22-32.
2015
- Doyle, T., Goujon, C., and Malim, M.H. (2015). HIV-1 and interferons: who’s interfering with whom? Nature Reviews Microbiology 13, 403-413.
- Goujon, C.#, Greenbury, R.A., Papaioannou, S., Doyle, T., and Malim, M.H.# (2015). A Triple-Arginine Motif in the Amino-Terminal Domain and Oligomerization Are Required for HIV-1 Inhibition by Human MX2. Journal of Virology 89, 4676-4680.
2014
- Goujon, C.#, Moncorge, O., Bauby, H., Doyle, T., Barclay, W.S., and Malim, M.H.# (2014). Transfer of the amino-terminal nuclear envelope targeting domain of human MX2 converts MX1 into an HIV-1 resistance factor. Journal of Virology 88, 9017-9026.
- Schaller, T., Bauby, H., Hué, S, Malim, MH, Goujon, C#. (2014) New insights into an X-traordinary viral protein. Frontiers in Microbiology. Apr 8;5:126.
2013
- Goujon, C., Moncorge, O., Bauby, H., Doyle, T., Ward, C.C., Schaller, T., Hue, S., Barclay, W.S., Schulz, R., and Malim, M.H. (2013). Human MX2 is an interferon-induced post-entry inhibitor of HIV-1 infection. Nature 502, 559-562.
- Goujon, C.*, Schaller, T.*, Galao, R.P., Amie, S.M., Kim, B., Olivieri, K., Neil, S.J., and Malim, M.H. (2013). Evidence for IFNalpha-induced, SAMHD1-independent inhibitors of early HIV-1 infection. Retrovirology 10, 23
2012
- Schaller, T., Goujon, C., and Malim, M.H. (2012). AIDS/HIV. HIV interplay with SAMHD1. Science 335, 1313-1314.
Local
- L. Chaloin. 3D model of MX proteins. Inhibitors of SARS-CoV-2.
- M. Blaise. Structure-function analysis of NCOA7, DDX42 and MX proteins.
- L. Espert, IRIM, Montpellier. Autophagy, innate responses and viruses
- J.L. Battini, IRIM and V. Courgnaud, IGMM, Montpellier. Genetic screens in haploid cells.
- L. Briant, IRIM, Montpellier. Identification of anti-arboviruses ISGs. Inhibition of CHIKV by DDX42.
- D. Muriaux & C. Favard, IRIM, Montpellier. MX interactions with lipids.
- L. Picas, IRIM, Montpellier. MX interactions with lipids.
- A. Bourdin, CHU de Montpellier. Epithéliums primaires respiratoires.
- J.M. Bellanger, CEFE. Antiviral activity of mushroom extracts.
- M. Moriel, CRBM. Inhibitors of SARS-CoV-2.
National
- Consortium ANRS MIE PEPR 3D-LUNGO
- M. Wencker, CIRI, Lyon. Potential modulation of immune responses by NCOA7 in mice.
- E. Ricci et P.E. Mangeot, CIRI, ENS Lyon. CRISPR/Cas9 genetic modification of primary cells using the Nanoblade techonology. Host responses to SARS-CoV-2.
- B. Reina San Martin, IGBMC, Strasbourg. Proteomics of HIV-1 reverse transcription complexes.
- E. Meunier and R. Planès, IPBS, Toulouse. Impact of NCOA7 on bacterial infections.
- C. Cougoule, IPBS, Toulouse. Organoid infection with SARS-CoV-2.
- S. Belouzard, Institut Pasteur de Lille. Impact sur MERS-CoV de gènes humains régulant SARS-CoV-2 et les coronavirus saisonniers.
International
- Consortium Horizon Europe APPEAL
- J. Doench, Broad Institute, USA. Genome-wide screens to identify SARS-CoV-2 regulators
- G. Kochs, Université de Freiburg, Allemagne. MX and innate immunity
- X. Saelens, Ghent university, Belgique. MX1 mode of action.
- M.H. Malim, King’s College London, Royaume-Uni. Early responses to HIV-1 infection.
- W.S. Barclay, Imperial College Lodon, Royaume-Uni. Study of influenza A virus.
- K. Peng, Research group of virus-host interactions, Wuhan Institute of Virology, Chinese Academy of Sciences. New dependency factors of HIV-1.
- J. Doench, Broad Institute, USA. CRISPR screens to identify SARS-CoV-2 regulators.
Septembre 2024 : L’équipe gagne ENFIN la pipette d’or aux journées IRIM

Avril 2024 : Antoine Rebendenne reçoit le prix de thèse de la Société Française de Virologie aux JFV, catégorie « virus émergents » 

Décembre 2023 : Antoine Rebendenne reçoit le Young Scientist Award du Pôle Biosanté de l’université de Montpellier, catégorie Infectiologie et Immunité pour son travail de thèse publié dans Nature Genetics. Bravo Antoine !
Novembre 2023 : Soutenance de doctorat d’Antoine Rebendenne sdevant un jury international. Bravo Antoine !
Novembre 2023 : Caroline Goujon est promue DR2 Inserm. Mai 2023 : Caroline Goujon a obtenu un financement ERC Consolidator Grant 2022 pour le projet InVIRium (Investigating Virus-Host Interplay in Human Primary Models of Genetically Modified Respiratory Epithelium) 
 Avril 2023 Joe McKellar reçoit le prix de thèse de la Société Française de Virologie aux JFV, catégorie « autres virus »
Avril 2023 Joe McKellar reçoit le prix de thèse de la Société Française de Virologie aux JFV, catégorie « autres virus »


Novembre 2022 : Publication dans Journal of Biological Chemistry du premier article premire auteur de Joe McKellar. An evolutionarily conserved N-terminal leucine is essential for MX1 GTPase antiviral activity against different families of RNA viruses.
17 Novembre 2022 : Soutenance de doctorat de Joe McKellar ! Félicitations, Joe !!



Une des images de microscopie de Joe McKellar selectionnée comme Image of dictinction at Nikon Small World 2022 Photomicrography competition
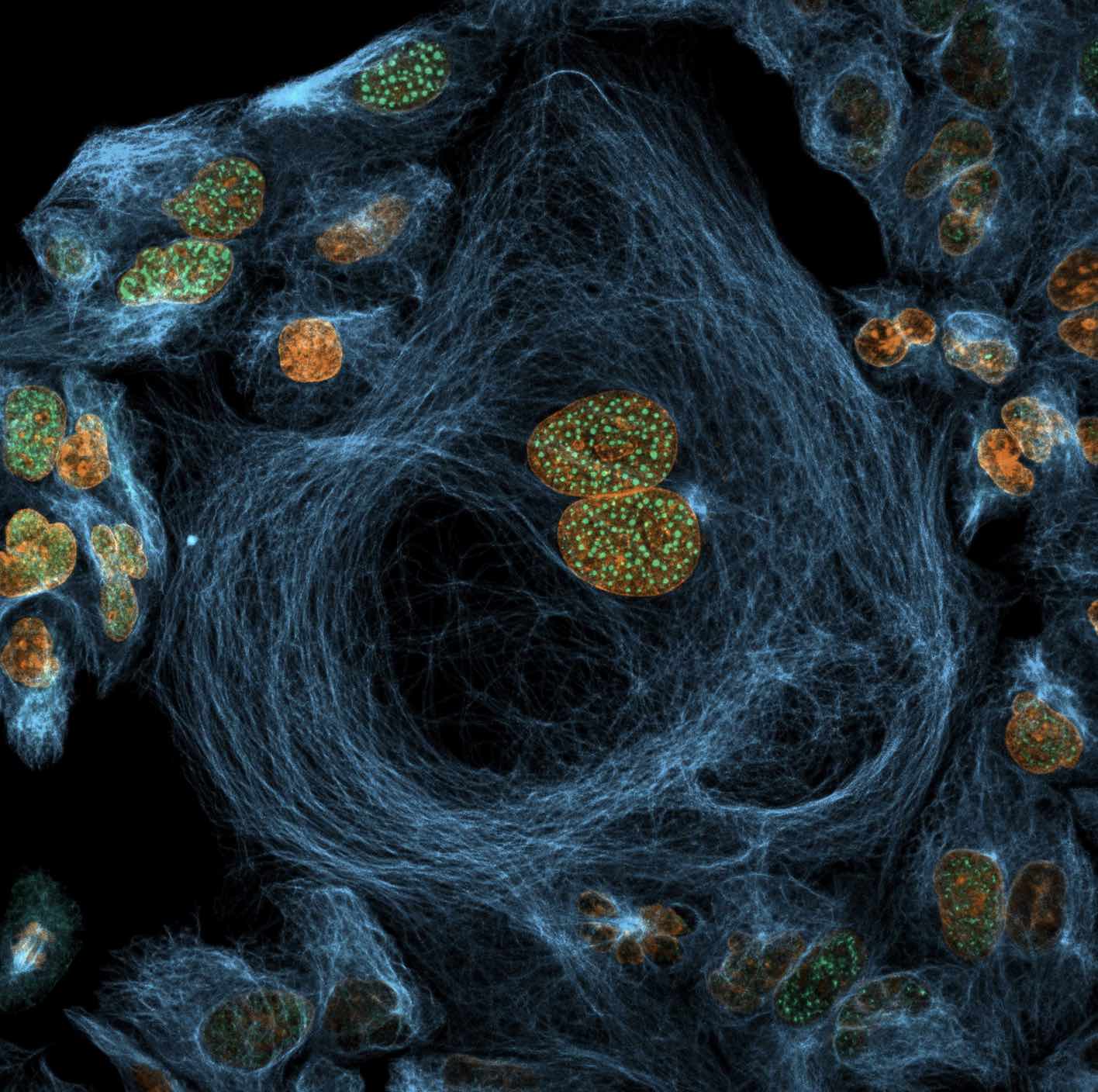
Août 2022 : La revue de notre ancien doctorant, Boris Bonaventure, sur les hélicases à ARN à la frontière de l’immunité intrinsèque et innée est en ligne : DExH/D-box helicases at the frontline of intrinsic and innate immunity against viral infections Juillet 2022 : Notre étude collaborative sur l’identification de gènes régulant la réplication du SARS-CoV-2 dans différentes lignées modèles est publiée chez Nature Genetics : Bidirectional genome-wide CRISPR screens reveal host factors regulating SARS-CoV-2, MERS-CoV and seasonal HCoVs
Avril 2022 : Nouveau preprint! The N-terminal domain of MX1 proteins is essential for their antiviral activity against different families of RNA viruses Octobre 2021 : Nouvelle courte revue de la littérature (forum) par C. Goujon, N. Jouvenet et A. Banerjee, publié dans Trends in Immunology Clash of the titans: interferons and SASR-CoV-2 Août 2021 : Notre étude dévoilant la structure tridimensionnelle du domaine TLDc de NCOA7-AS, menée par notre collaborateur structuraliste Mickaël Blaise, est en ligne ! Crystal structure of the TLDc domain of human NCOA7-AS Juin 2021. Joe McKellar obtient une 4e année de financement de thèse de La Fondation pour la Recherche Médicale (FRM).

Mai 2021 : Second preprint de l’équipe sur le SARS-CoV-2 ! En collaboration avec John Doench (Broad Institute, USA), nous avons réalisé des cribles CRISPR bidirectionnels à l’échelle du génome dans des cellules relevantes physiologiquement et exprimant naturellement ACE2 et TMPRSS2. Nous avons ainsi identifié de nouveaux gènes de dépendance du SARS-CoV-2 et de nouveaux inhibiteurs ! Certains de ces gènes régulent aussi la réplication du MERS-CoV (résultats obtenus en collaboration avec l’équipe de Jean Dubuisson, Institut Pasteur Lille), et des coronavirus saisonniers HCoV-NL63 et HCoV-229E. A lire ici : https://www.biorxiv.org/content/10.1101/2021.05.19.444823v2
Janvier 2021 : la première publication de l’équipe sur le SARS-CoV-2 accepté par Journal of Virology ! https://jvi.asm.org/content/early/2021/01/28/JVI.02415-20 Dans cette étude, nous montrons une très forte réponse interféron dans les épithéliums respiratoires primaires et des lignées modèles, via le senseur MDA-5. Mais cette réponse interféron ne permet pas le contrôle de la réplication, car elle intervient trop tardivement.
Décembre 2020 : Reportage sur l’équipe dans le magazine Inserm magazine, également accessible sur le site web de l’inserm : https://fr.calameo.com/read/005154450ae7eeb3925f0 https://www.inserm.fr/en/research-inserm/live-from-our-labs/laboratory-in-motion/sars-cov-2-under-microscopes

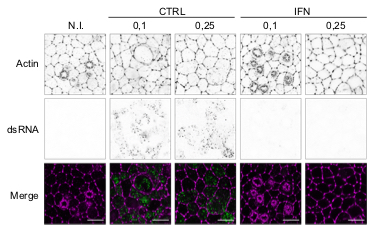
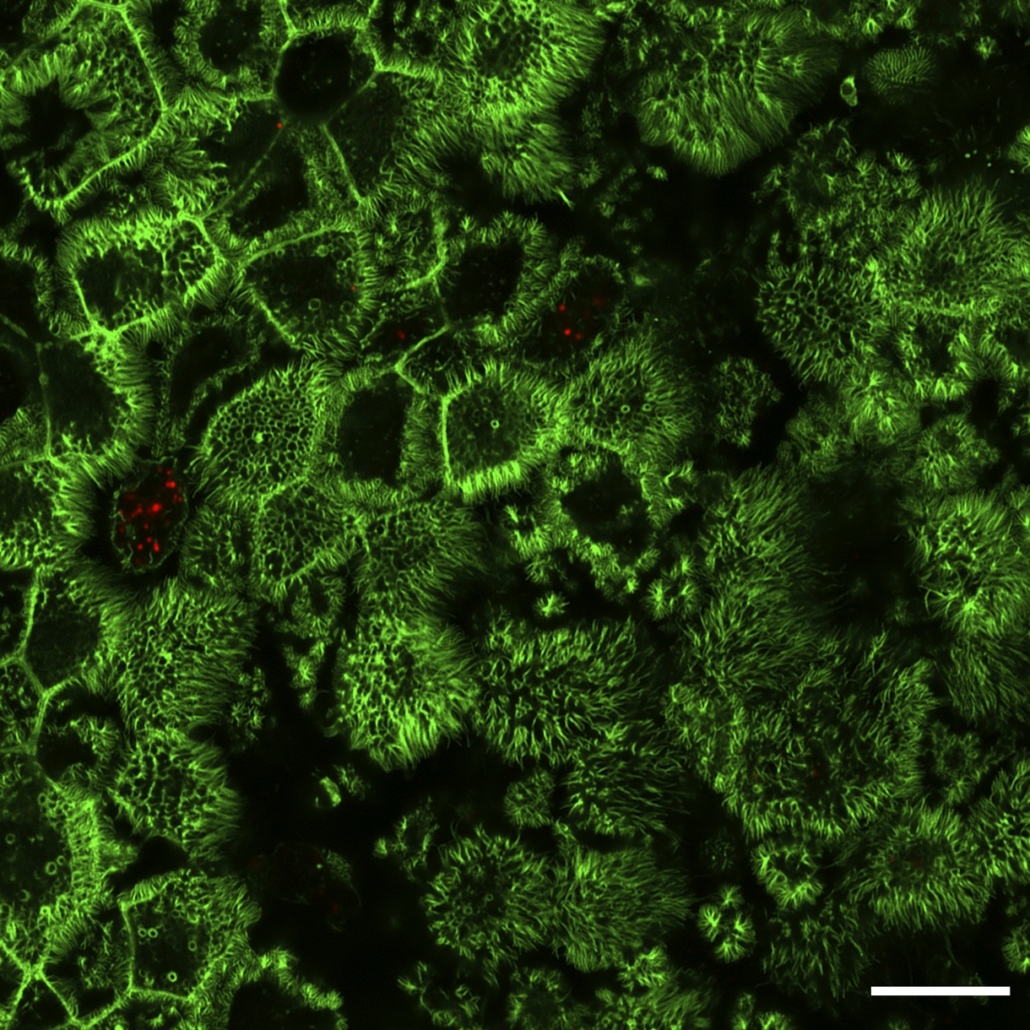
Octobre 2020 : les deux premiers preprints de l’équipe sont en ligne ! The first preprint describes the identification of DDX42 as a broad antiviral inhibitor. Boris Bonaventure, in the team, performed whole-genome CRISPR screens which led to the identification of DDX42 as an intrinsic inhibitor of HIV-1, in various cell lines and primary cells. DDX42 inhibits viral DNA accumulation and is in close proximity with Capsid in infected cells. DDX42 is also capable of inhibiting retrotranspoition of Line-1 and infection with different retroviruses. Finally, we reveal that DDX42 is also a potent inhibitor of other pathogenic viruses, including SARS-CoV-2! https://www.biorxiv.org/content/10.1101/2020.10.28.359356v1 All the team members participated in this work as well as Laurence Briant and Eric Bernard from IRIM (work on CHIKV) as well as Nathalie Gros from CEMIPAI (work on ZIKV). The library preparation and deep sequencing analysis was done by Hugues Parrinello and Stéphanie Rialle from Montpellier GenomiX. In the second preprint, we have explored the host cell responses to SARS-CoV-2 both in primary human airway epithelia and in model cell lines. We observed a potent interferon induction, mainly due to sensing by MDA-5, but unable to control replication. https://www.biorxiv.org/content/10.1101/2020.10.28.358945v1 This work was done in collaboration with cytokine specialists, Seb Nisole and Ghizlane Maarifi at the IRIM. Ci-dessous un exemple d’image prise au microscope LSM880 en mode Airyscan de cellules primaires respiratoires humaines (en vert, Actine) infectées par SARS-CoV-2 (rouge). Admirez les cils sur cette vue de dessus ! Barre d’échelle : 10 um, IF et image réalisées par Joe McKellar, infection par Olivier Moncorgé.
Septembre 2020 : Obtention par C. Goujon des deux premiers financements ANR de l’équipe ! ANR RA-COVID pour le projet CRISPR-TARGET-CoV visant à identifier de nouvelles cibles thérapeutiques pour le SARS-CoV-2 par cribles CRISPR à l’échelle du génome, en collaboration avec John Doench (Broad Institute, USA). ANR PRC pour le projet CAIPIRINAS (Control of type A Influenza virus Propagation and Immune Responses: Investigating the role of NCOA7-Alternative Start), en collaboration avec M. Blaise et M. Wencker. Ce projet vise à étudier le mode d’action in vitro, in cellulo et le rôle in vivo de NCOA7.

Juin 2020 : Antoine Rebendenne, normalien agrégé de l’ENS-Paris Saclay, reçoit un financement de thèse pour effectuer sa thèse dans l’équipe. L’inserm consacre une page web aux travaux de l’équipe sur le SARS-CoV-2 : https://www.inserm.fr/actualites-et-evenements/actualites/quand-covid-19-reoriente-travaux-equipe-virologues Printemps 2020 : Obtention de financements auprès de MUSE, de l’INSB et la Fondation CNRS pour les travaux de l’équipe sur le SARS-CoV-2. 


Mars 2020 : Alors que la France est en confinement généralisé, des membres de l’IRIM dont notre équipe se mobilisent pour la recherche sur le SARS-CoV-2, virus responsable de la pandémie mondiale de covid19. Dans l’équipe, C. Goujon, O. Moncorgé et M. Tauziet sont au laboratoire pour réaliser des expériences, avec un support à distance des autres membres de l’équipe, en particulier de B. Bonaventure. A. Chaves Valadao renforce l’équipe COVID-19 en Avril 2020, puis A. Rebendenne dès la fin du premier confinement, à la mi-mai.

Mars-Avril 2020 : Les membres de l’équipe mobilisés pour la recherche contre le covid19. De gauche à droite : Marine Tauziet dans le labo de biologie moléculaire, Olivier Moncorgé au P2, au P3 (CEMIPAI), Caroline Goujon au P3 Janvier 2020 : un financement ERC Proof of Concept accordé à C. Goujon sur le 3e call de 2019 pour le projet FluAttack mené par O. Moncorgé (ERC website)


Août 2019: Obtention de deux financements pré-maturation de la région Occitanie : pour le projet Antigrip Anticip mené par Olivier Moncorgé dans l’équipe et le projet Fongal Kombat Microbes, porté par Jean-Michel Bellanger du CEFE en collaboration avec l’équipe de Matteo Bonazzi et notre équipe.


Septembre 2019 : Apéritif à l’IRIM pour lêter les financements de la Région.
June 2019. Boris Bonaventure reçoit une 4e année de financement de thèse de la part de La Fondation pour la Recherche Médicale (FRM). 
Mars 2019. Six membres de l’équipe aux Journées Francophones de Virologie à l’ENS de Lyon et de nombreux représentants de l’IRIM ! Et prix du second meilleur poster pour Boris Bonaventure : bravo, Boris !


Novembre 2018. Publication dans Nature Microbiology de notre article collaboratif avec l’équipe de M. Malim au King’s College London décrivant un nouvel ISG antiviral: Doyle T., Moncorgé O., Bonaventure B., Pollpeter D., Lussignol M., Tauziet M., Apolonia L., Catanese M.T., Goujon C.# and Malim M.H.#. The interferon inducible isoform of NCOA7 inhibits endosome-mediated viral entry. Nature Microbiology, (12):1369-1376. (# corresponding authors)
Lien vers le post ‘Behind the paper’ de C. Goujon : Entry forbidden, a new antiviral ISG identified
Juin 2018. Joe McKellar, en Master 2 dans l’équipe, classé 1er au concours de l’école doctorale CBS2 pour l’obtention de la bourse de thèse (MRT). Mars 2018. Article sur Caroline Goujon sur le site web de l’INSERM
Septembre 2017 : Départ de Rémi Planès. Après 2 ans de post-doctorat dans l’équipe, Rémi nous quitte pour retourner à Toulouse et y continuer sa carrière scientifique, sur l’inflammasome. Bonne chance pour la suite Rémi !


Septembre 2017 : Apéro-déjeunatoire à l’IRIM pour le départ de Rémi.
Juillet 2017 : Caroline Goujon a obtenu un financement ERC Starting Grant pour le projet ANTIViR (Mechanisms of interferon-induced antiviral restriction and signalling)




Juillet 2017 : Apéro-déjeunatoire à l’IRIM pour célébrer les financements obtenus.
Janvier 2017: Olivier Moncorgé a obtenu un renouvellement de 1 an de son financement post-doctoral auprès de l’ANRS, pour la caractérisation de facteurs cellulaires identifiés par un crible génétique CRISPR/Cas9 inhibant le VIH-1. 
Juillet 2016 : Caroline Goujon a obtenu en collaboration avec Jean-Luc Battini (IRIM, ex-IGMM) et Valérie Courgnaud (IGMM) des financements Sidaction et ANRS de 2 et 3 ans, respectivement, pour l’identification des cofacteurs de MX2 et d’autres inhibiteurs du VIH-1 induits par l’interféron de type 1.


Juin 2016 : Boris Bonaventure, en Master 2 dans l’équipe, classé 2e au concours de l’école doctorale CBS2 pour l’obtention de la bourse de thèse (MRT). Juillet 2015 : Rémi Planès a obtenu un financement postdoctoral de 2 ans auprès de Sidaction pour rejoindre l’équipe et étudier le rôle potentiel de MX2 dans la signalisation de l’immunité innée.


Opened: 2 Postdoctoral positions on innate immunity and respiratory viruses from 2024
We are recruiting 2 postdoctoral fellows on innate immunity and respiratory viruses to work on the ERC-funded InVIRium project, for up to 4 and 5 years, respectively:
– 1 postdoctoral fellow with a strong expertise in the fields of innate immunity and virology (experience in BSL-3 lab, and with respiratory viruses required). Experience in 3D-cultures of primary airway epithelia would be a massive plus.
– 1 postdoctoral fellow with skills in bioinformatics (scRNA-seq and NGS analyses) and in wet lab experiments, with expertise in molecular virology.
The InVIRium project aims at understanding the relationships between respiratory viruses and their host cells, using highly pertinent models of airway epithelia cultured at the air-liquid interface, with a major focus on respiratory viruses (coronaviruses, influenza viruses and respiratory syncytial virus) and on interferon-induced antiviral restriction.
The IRIM offers access to all the facilities required for cutting-edge research, including BSL2 and BSL3 labs, state-of-the-art cell imaging and microscopy facilities, flow cytometry and cell sorting, genomics, and proteomics platforms. The IRIM constitutes a friendly and international working environment, with staff and students coming from around 25 different countries. Montpellier is a lovely city in the South of France, 15 km away from the Mediterranean sea and offering an amazing quality of life. The 2 positions will be available from the beginning of 2024.
Candidates should send their application (CV and motivation, plus contact for 2-3 referees) by email to Caroline Goujon
En bref
L’état antiviral induit par l’interféron inhibe fortement la réplication virale dans les cellules humaines, ce qui signifie que celles-ci sont capables d’exprimer naturellement de puissants inhibiteurs antiviraux. L’équipe s’intéresse à ces mécanismes de défenses antiviraux induits par l’interféron. A l’aide d’approches puissantes, tels que des cribles génétiques fonctionnels à l’échelle du génome humain par knock-out CRISPR/Cas9, nous souhaitons identifier de nouveaux effecteurs de l’état antiviral puis comprendre leur activité antivirale.
Financements

Financements passés :




