2025
Structural basis of phosphate export by human XPR1. He Q, Zhang R, Tury S, Courgnaud V, Liu F, Battini JL, Li B, Chen Q. Nat Commun. 2025 Jan 15;16(1):683. doi: 10.1038/s41467-025-55995-8. PMID: 39814721
2024
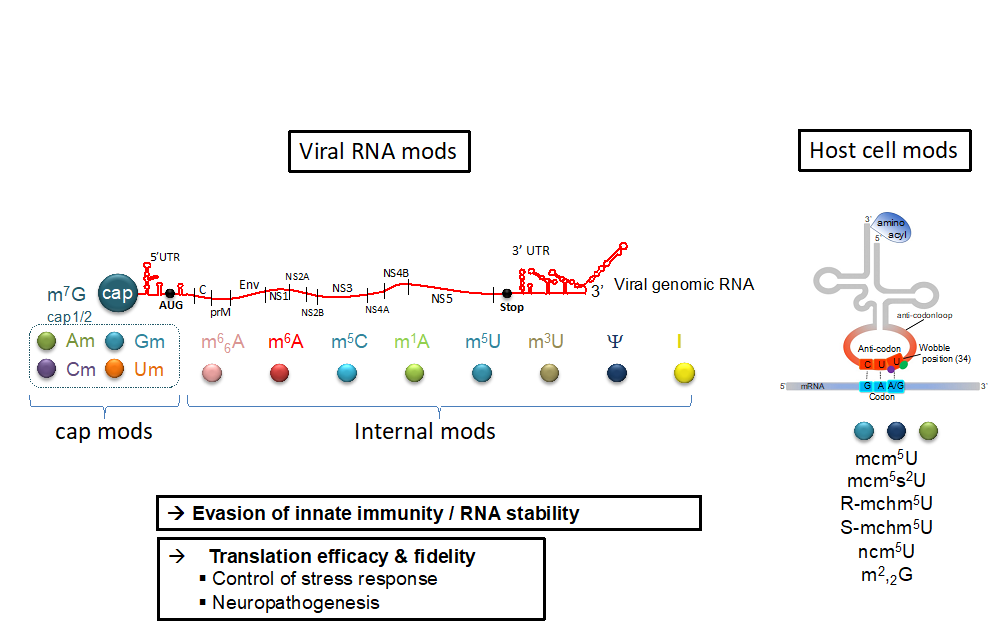
SARS-CoV-2 Displays a Suboptimal Codon Usage Bias for Efficient Translation in Human Cells Diverted by Hijacking the tRNA Epitranscriptome. Eldin P, David A, Hirtz C, Battini JL, Briant L. Int J Mol Sci. 2024 Oct 29;25(21):11614. doi: 10.3390/ijms252111614. PMID: 39519170
The Rac1-PAK1-Arp2/3 signaling axis regulates CHIKV nsP1-induced filopodia and optimal viral genome replication. Aïqui-Reboul-Paviet O, Bakhache W, Bernard E, Holsteyn L, Neyret A, Briant L. J Virol. 2024 Oct 22;98(10):e0061224. doi: 10.1128/jvi.00612-24. Epub 2024 Sep 19. PMID: 39297643
In situ fate of Chikungunya virus replication organelles. Girard J, Le Bihan O, Lai-Kee-Him J, Girleanu M, Bernard E, Castellarin C, Chee M, Neyret A, Spehner D, Holy X, Favier A-L, Briant L, Bron P. J Virol. 2024 Jul 23;98(7):e0036824. doi: 10.1128/jvi.00368-24. Epub 2024 Jun 28. PMID: 38940586
Proteolytic cleavage and inactivation of the TRMT1 tRNA modification enzyme by SARS-CoV-2 main protease. Zhang K, Eldin P, Ciesla JH, Briant L, Lentini JM, Ramos J, Cobb J, Munger J, Fu D. Elife. 2024 May 30;12:RP90316. doi: 10.7554/eLife.90316. PMID: 38814682
Biallelic NAA60 variants with impaired n-terminal acetylation capacity cause autosomal recessive primary familial brain calcifications. Chelban V, Aksnes H, Maroofian R, LaMonica LC, Seabra L, Siggervåg A, Devic P, Shamseldin HE, Vandrovcova J, Murphy D, Richard AC, Quenez O, Bonnevalle A, Zanetti MN, Kaiyrzhanov R, Salpietro V, Efthymiou S, Schottlaender LV, Morsy H, Scardamaglia A, Tariq A, Pagnamenta AT, Pennavaria A, Krogstad LS, Bekkelund ÅK, Caiella A, Glomnes N, Brønstad KM, Tury S, Moreno De Luca A, Boland-Auge A, Olaso R, Deleuze JF, Anheim M, Cretin B, Vona B, Alajlan F, Abdulwahab F, Battini JL, İpek R, Bauer P, Zifarelli G, Gungor S, Kurul SH, Lochmuller H, Da’as SI, Fakhro KA, Gómez-Pascual A, Botía JA, Wood NW, Horvath R, Ernst AM, Rothman JE, McEntagart M, Crow YJ, Alkuraya FS, Nicolas G; SYNaPS Study Group; Arnesen T, Houlden H. Nat Commun. 2024 Mar 13;15(1):2269. doi: 10.1038/s41467-024-46354-0. PMID: 38480682
2023
A co-opted endogenous retroviral envelope promotes cell survival by controlling CTR1-mediated copper transport and homeostasis. Tury S, Chauveau L, Lecante A, Courgnaud V, Battini JL. Cell Rep. 2023 Sep 26;42(9):113065. doi: 10.1016/j.celrep.2023.113065. Epub 2023 Sep 7. PMID: 37682705
Editorial: Broadening our view on nucleic acid sensing: novel sensors, signaling pathways, and involvement in non-infectious diseases. Chauveau L, Laguette N, Sampaio NG. Front Immunol. 2023 Aug 11;14:1266732. doi: 10.3389/fimmu.2023.1266732. eCollection 2023. PMID: 37638050
cGAMP the travelling messenger. Blest HTW, Chauveau L. Front Immunol. 2023 May 23;14:1150705. doi: 10.3389/fimmu.2023.1150705. eCollection 2023. PMID: 37287967
2022
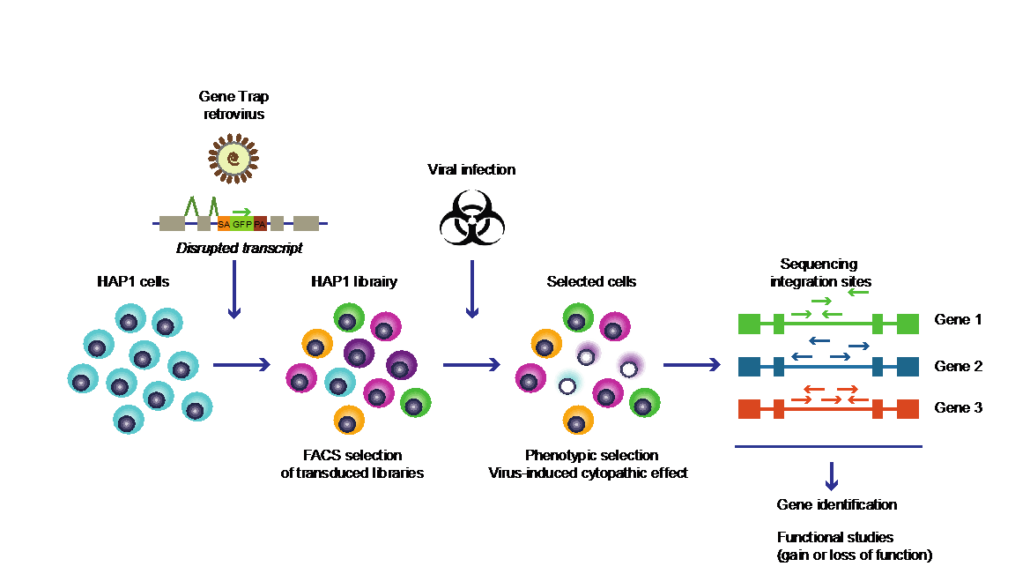
The DEAD box RNA helicase DDX42 is an intrinsic inhibitor of positive-strand RNA viruses. Bonaventure B, Rebendenne A, Chaves Valadão AL, Arnaud-Arnould M, Gracias S, Garcia de Gracia F, McKellar J, Labaronne E, Tauziet M, Vivet-Boudou V, Bernard E, Briant L, Gros N, Djilli W, Courgnaud V, Parrinello H, Rialle S, Blaise M, Lacroix L, Lavigne M, Paillart JC, Ricci EP, Schulz R, Jouvenet N, Moncorgé O, Goujon C. EMBO Rep. 2022 Nov 7;23(11):e54061. doi: 10.15252/embr.202154061. Epub 2022 Sep 26. PMID: 36161446
Identification of a non-canonical G3BP-binding sequence in a Mayaro virus nsP3 hypervariable domain. Neyret A, Bernard E, Aïqui-Reboul-Paviet O, Bakhache W, Eldin P, Chaloin L, Briant L. Front Cell Infect Microbiol. 2022 Aug 11;12:958176. doi: 10.3389/fcimb.2022.958176. eCollection 2022. PMID: 36034716
Identification of Copper Transporter 1 as a Receptor for Feline Endogenous Retrovirus ERV-DC14. Tury S, Giovannini D, Ivanova S, Touhami J, Courgnaud V, Battini JL. J Virol. 2022 Jun 22;96(12):e0022922. doi: 10.1128/jvi.00229-22. Epub 2022 Jun 2. PMID: 35652657
2021
New Insights into Chikungunya Virus Infection and Pathogenesis. Kril V, Aïqui-Reboul-Paviet O, Briant L, Amara A. Annu Rev Virol. 2021 Sep 29;8(1):327-347. doi: 10.1146/annurev-virology-091919-102021. Epub 2021 Jul 13. PMID: 34255544
2020
Impact of HIV-1 Vpr manipulation of the DNA repair enzyme UNG2 on B lymphocyte class switch recombination. Eldin P, Péron S, Galashevskaya A, Denis-Lagache N, Cogné M, Slupphaug G, Briant L. J Transl Med. 2020 Aug 10;18(1):310. doi: 10.1186/s12967-020-02478-7. PMID: 32778120
Neurocognitive impacts of arbovirus infections. Clé M, Eldin P, Briant L, Lannuzel A, Simonin Y, Van de Perre P, Cabié A, Salinas S. J Neuroinflammation. 2020 Aug 10;17(1):233. doi: 10.1186/s12974-020-01904-3. PMID: 32778106
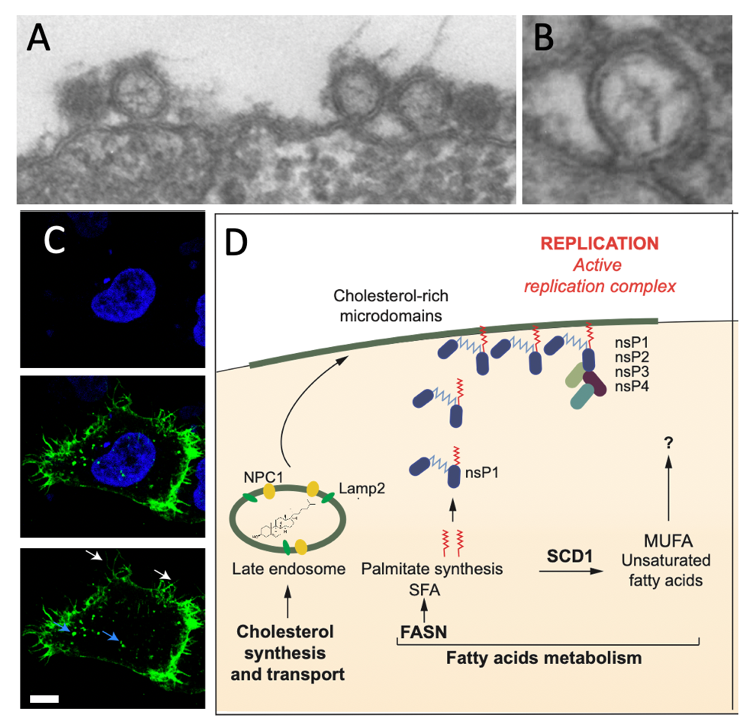
Palmitoylated Cysteines in Chikungunya Virus nsP1 Are Critical for Targeting to Cholesterol-Rich Plasma Membrane Microdomains with Functional Consequences for Viral Genome Replication. Bakhache W, Neyret A, Bernard E, Merits A, Briant L. J Virol. 2020 May 4;94(10):e02183-19. doi: 10.1128/JVI.02183-19. Print 2020 May 4. PMID: 32132240
Haploinsufficiency of the Primary Familial Brain Calcification Gene SLC20A2 Mediated by Disruption of a Regulatory Element. Cassinari K, Rovelet-Lecrux A, Tury S, Quenez O, Richard AC, Charbonnier C, Olaso R, Boland A, Deleuze JF, Besancenot JF, Delpont B, Pouliquen D, Lecoquierre F, Chambon P, Thauvin-Robinet C, Campion D, Frebourg T, Battini JL, Nicolas G. Mov Disord. 2020 Aug;35(8):1336-1345. doi: 10.1002/mds.28090. Epub 2020 Jun 7. PMID: 32506582
Interplay between primary familial brain calcification-associated SLC20A2 and XPR1 phosphate transporters requires inositol polyphosphates for control of cellular phosphate homeostasis. López-Sánchez U, Tury S, Nicolas G, Wilson MS, Jurici S, Ayrignac X, Courgnaud V, Saiardi A, Sitbon M, Battini JL. J Biol Chem. 2020 Jul 10;295(28):9366-9378. doi: 10.1074/jbc.RA119.011376. Epub 2020 May 11. PMID: 32393577
2019
Fatty acid synthase and stearoyl-CoA desaturase-1 are conserved druggable cofactors of Old World Alphavirus genome replication. Bakhache W, Neyret A, McKellar J, Clop C, Bernard E, Weger-Lucarelli J, Briant L. Antiviral Res. 2019 Dec;172:104642. doi: 10.1016/j.antiviral.2019.104642. Epub 2019 Oct 31. PMID: 31678479
[Architecture and biogenesis of positive-stranded RNA virus replication organelles]. Bakhache W, Couderc É, Neyret A, Briant L. Virologie (Montrouge). 2019 Jun 1;23(3):160-175. doi: 10.1684/vir.2019.0779. PMID: 31210133
Insight into the mechanism of action of EP-39, a bevirimat derivative that inhibits HIV-1 maturation. Neyret A, Gay B, Cransac A, Briant L, Coric P, Turcaud S, Laugâa P, Bouaziz S, Chazal N. Antiviral Res. 2019 Apr;164:162-175. doi: 10.1016/j.antiviral.2019.02.014. Epub 2019 Feb 27. PMID: 30825471
The Host DHX9 DExH-Box Helicase Is Recruited to Chikungunya Virus Replication Complexes for Optimal Genomic RNA Translation. Matkovic R, Bernard E, Fontanel S, Eldin P, Chazal N, Hassan Hersi D, Merits A, Péloponèse JM Jr, Briant L. J Virol. 2019 Feb 5;93(4):e01764-18. doi: 10.1128/JVI.01764-18. Print 2019 Feb 15. PMID: 30463980
2017
Incidence of dengue and chikungunya viruses in mosquitoes and human patients in border provinces of Vietnam. Pham Thi KL, Briant L, Gavotte L, Labbe P, Perriat-Sanguinet M, Cornillot E, Vu TD, Nguyen TY, Tran VP, Nguyen VS, Devaux C, Afelt A, Tran CC, Phan TN, Tran ND, Frutos R. Parasit Vectors. 2017 Nov 9;10(1):556. doi: 10.1186/s13071-017-2422-z. PMID: 29121985
Peptides derived from evolutionarily conserved domains in Beclin-1 and Beclin-2 enhance the entry of lentiviral vectors into human cells. Majdoul S, Cosette J, Seye AK, Bernard E, Frin S, Holic N, Chazal N, Briant L, Espert L, Galy A, Fenard D. J Biol Chem. 2017 Nov 10;292(45):18672-18681. doi: 10.1074/jbc.M117.800813. Epub 2017 Sep 19. PMID: 28928217
Imipramine Inhibits Chikungunya Virus Replication in Human Skin Fibroblasts through Interference with Intracellular Cholesterol Trafficking. Wichit S, Hamel R, Bernard E, Talignani L, Diop F, Ferraris P, Liegeois F, Ekchariyawat P, Luplertlop N, Surasombatpattana P, Thomas F, Merits A, Choumet V, Roques P, Yssel H, Briant L, Missé D. Sci Rep. 2017 Jun 9;7(1):3145. doi: 10.1038/s41598-017-03316-5. PMID: 28600536
2016
Zika Virus Strains Potentially Display Different Infectious Profiles in Human Neural Cells. Simonin Y, Loustalot F, Desmetz C, Foulongne V, Constant O, Fournier-Wirth C, Leon F, Molès JP, Goubaud A, Lemaitre JM, Maquart M, Leparc-Goffart I, Briant L, Nagot N, Van de Perre P, Salinas S. EBioMedicine. 2016 Oct;12:161-169. doi: 10.1016/j.ebiom.2016.09.020. Epub 2016 Sep 21. PMID: 27688094
[Zika virus, an emerging threat]. Salinas S, Foulongne V, Loustalot F, Fournier-Wirth C, Molès JP, Briant L, Nagot N, Van de Perre P, Simonin Y. Med Sci (Paris). 2016 Apr;32(4):378-86. doi: 10.1051/medsci/20163204016. Epub 2016 May 2. PMID: 27137695
Surveillance of dengue and chikungunya infection in Dong Thap, Vietnam: A 13-month study. Kim Lien PT, Briant L, Tang TB, Trang BM, Gavotte L, Cornillot E, Duoc VT, Duong TN, Frutos R, Nga PT. Asian Pac J Trop Med. 2016 Jan;9(1):39-43. doi: 10.1016/j.apjtm.2015.12.008. Epub 2015 Dec 19. PMID: 26851784
2015
Role of Aedes aegypti and Aedes albopictus during the 2011 dengue fever epidemics in Hanoi, Vietnam. Kim Lien PT, Duoc VT, Gavotte L, Cornillot E, Nga PT, Briant L, Frutos R, Duong TN. Asian Pac J Trop Med. 2015 Jul;8(7):543-8. doi: 10.1016/j.apjtm.2015.06.009. Epub 2015 Jul 9. PMID: 26276285
Biology of Zika Virus Infection in Human Skin Cells. Hamel R, Dejarnac O, Wichit S, Ekchariyawat P, Neyret A, Luplertlop N, Perera-Lecoin M, Surasombatpattana P, Talignani L, Thomas F, Cao-Lormeau VM, Choumet V, Briant L, Desprès P, Amara A, Yssel H, Missé D. J Virol. 2015 Sep;89(17):8880-96. doi: 10.1128/JVI.00354-15. Epub 2015 Jun 17. PMID: 26085147
Inflammasome signaling pathways exert antiviral effect against Chikungunya virus in human dermal fibroblasts. Ekchariyawat P, Hamel R, Bernard E, Wichit S, Surasombatpattana P, Talignani L, Thomas F, Choumet V, Yssel H, Desprès P, Briant L, Missé D. Infect Genet Evol. 2015 Jun;32:401-8. doi: 10.1016/j.meegid.2015.03.025. Epub 2015 Apr 4. PMID: 25847693
Montpellier Infectious Diseases – Pôle Rabelais (MID) 3rd annual meeting (2014). Besteiro S, Blanc-Potard A, Bonazzi M, Briant L, Chazal N, Cornillot E, Lentini G, Matkovic R, Sanosyan A, Tuaillon E, Van de Perre P. Infect Genet Evol. 2015 Jun;32:161-4. doi: 10.1016/j.meegid.2015.03.007. Epub 2015 Mar 16. PMID: 25791932
Human keratinocytes restrict chikungunya virus replication at a post-fusion step. Bernard E, Hamel R, Neyret A, Ekchariyawat P, Molès JP, Simmons G, Chazal N, Desprès P, Missé D, Briant L. Virology. 2015 Feb;476:1-10. doi: 10.1016/j.virol.2014.11.013. Epub 2014 Dec 8. PMID: 25496825