Notre équipe s’intéresse au rôle des protéines virales, et cellulaires, et de leur interactions avec l’ARN viral et des lipides de la cellule hôte au cours de la traduction virale et dans l’assemblage des virus. Nous étudions ces phénomènes de façon quantitative à l’échelle de la molécule unique dans des systèmes modèles, dans des cellules hôtes et organismes multi-cellulaires vivants.
Nos axes de recherches comportent 6 grandes questions :
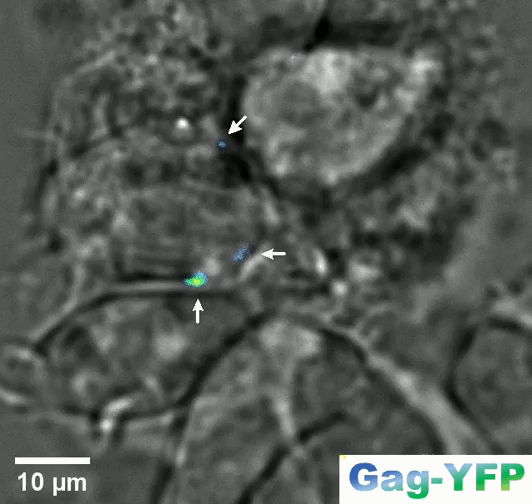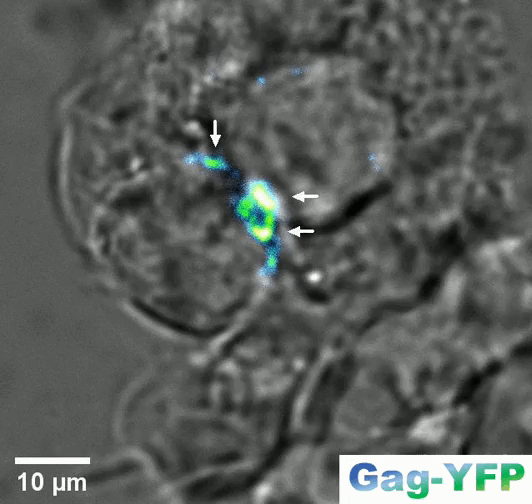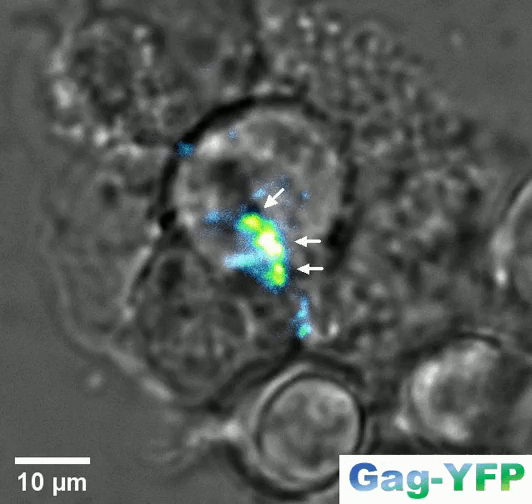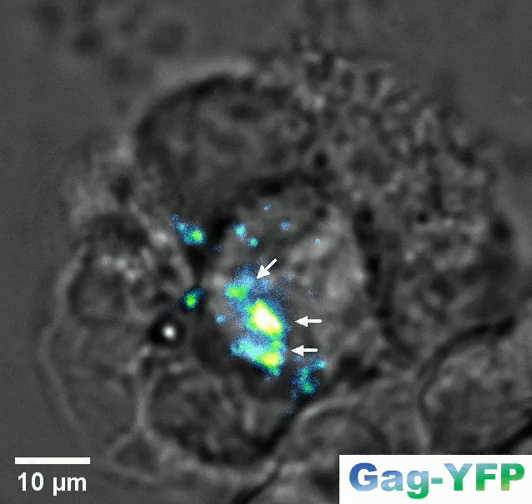
Retroviral biofilm assembly and polarization at the T cell-T cell junction (from Arone et al., mBio 2023)
- Tri des lipides lors de l’auto-assemblage des protéines Gag du HIV-1 et M1 du virus de la grippe A.(Kerviel 2013; Saad & Muriaux, 2015; Kerviel 2016; Yandrapalli 2015, 2016; Favard 2019)
- Microscopie de molécule unique de l’assemblage de HIV-1 dans des lymphocytes T CD4 vivants. (Mariani 2016; Mariani 2018; Inamdar 2019; Dibsy 2023)
- Dynamique de l’actine corticale et courbure membranaire au cours de l’assemblage viral. (Thomas 2015; Inamdar 2021; Dibsy 2023)
- Rôle des protéines de matrice M et de cofacteurs cellulaires dans l’assemblage du virus de la grippe et du SARS-CoV-2. (Kerviel 2016; Bracquemond & Muriaux, 2021; Gourdelier 2023; Swain, 2023; Swain, 2023)
- Etude par microscopie à force atomique (AFM) de l’assemblage du HIV-1, IAV et SARS-CoV-2 sur des particules virales uniques. (Faivre-Moskalenko 2014; Bernaud 2015; Lyonnais 2021)
- Etude de la dynamique de réplication virale (traduction/amplification) en cellules et au cours du développement. (Dufourt et al., Science 2022; Bellec et al., Nat Comm 2022; Bellec et al., BioRXiV 2023)
1. Tri des lipides lors de l’auto-assemblage des protéines Gag du HIV-1 et M1 du virus de la grippe A
(Financement : ANR Fluobuds, CNRS, Univ Montp, GdR IMABIO, ERC Erasmus Mundus) Ce projet vise à déchiffrer le rôle de nanodomaines lipidiques préexistant ou générés dans la membrane plasmique de la cellule hôte lors de l’assemblage viral. Lors de précédentes études, nous avons montré que l’interaction entre le domaine Matrice de la protéine Gag et des lipides spécifiques de la membrane plasmique (PS/PIP2) induit l’assemblage des rétrovirus (Hamard-Péron et al., 2010 ; Hamard-Péron & Muriaux, 2011). Nous avons proposé que des microdomaines enrichis en lipides acides (ALEM) sont crées par l’oligomérisation de la protéine Gag de HIV-1 dans la membrane plasmique de la cellule hôte lors de l’assemblage viral (Kerviel et al., 2013, Yandapalli et al., 2014, Mariani et al., 2014 & Saad & Muriaux, Editorial 2015). Pour cela, nous avons caractérisé la nature et les effets de l’interaction du domaine MA des protéines Gag rétrovirales (HIV-1 et MLV) avec des membranes lipidiques in silico (Kerviel et al., 2013, Charlier et al., 2014). In vitro, nous nous sommes intéressés à l’impact de l’auto-assemblage des protéines Gag de HIV-1 sur la réorganisation de la membrane plasmique à l’aide de membranes modèles de composition moléculaires mimant cette membrane. Nous avons ainsi pu montrer que lors de son auto-assemblage, la protéine virale Gag trie les lipides membranaires et génére des nano-domaines lipidique enrichis en PIP2 et Cholestérol, tout en excluant la Sphingomyéline (Yandrapalli et al., 2016).
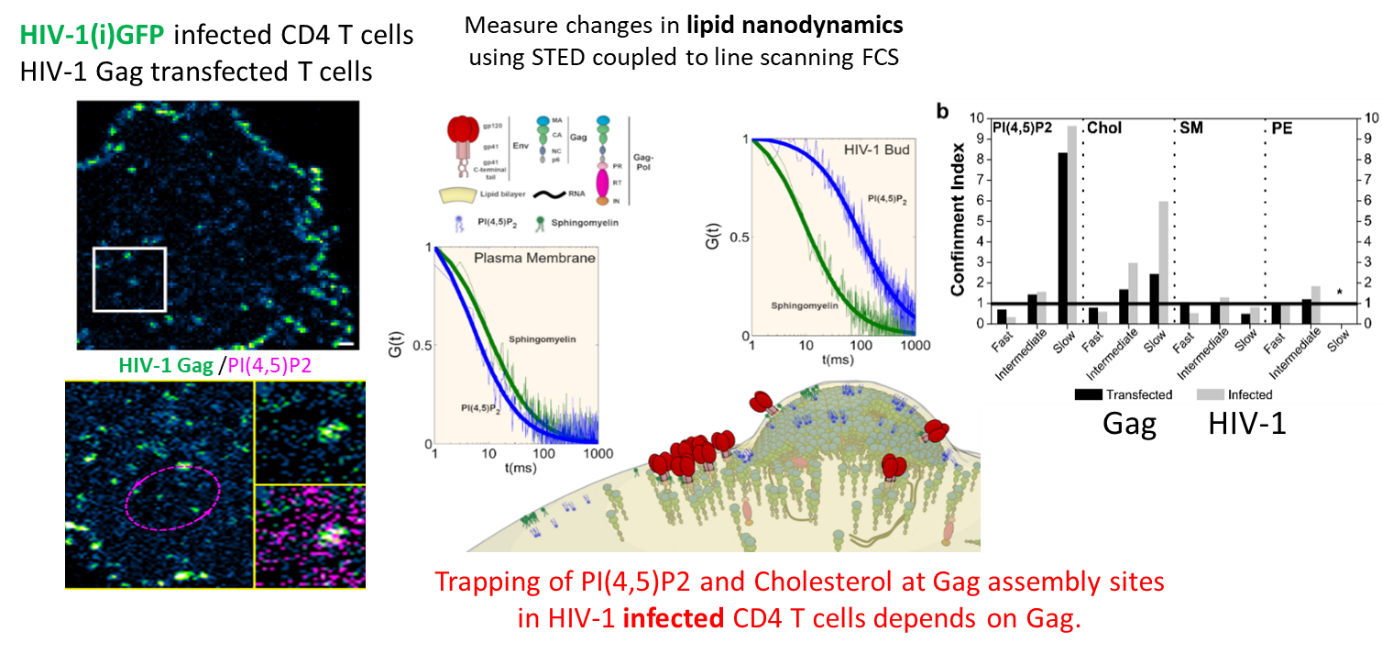 Nous avons finalement quantifié ce phénomène dans les cellules hôtes du virus (lymphocytes T CD4 infectés par HIV-1 ou transfectés par Gag) en mesurant les changement de diffusion des lipides (PIP2, Cholesterol, SM et PE) dans et en dehors du bourgeon viral à la surface des cellules T par sSTED-FCS (Scanning STED FCS), en collaboration avec C. Eggeling (U. Oxford/ Univ Jena, UK-Allemagne) et J Chojnacki (U. Oxford/ IRSI Caixa, Barcelona, SP) (Favard et al., 2019).
Nous avons finalement quantifié ce phénomène dans les cellules hôtes du virus (lymphocytes T CD4 infectés par HIV-1 ou transfectés par Gag) en mesurant les changement de diffusion des lipides (PIP2, Cholesterol, SM et PE) dans et en dehors du bourgeon viral à la surface des cellules T par sSTED-FCS (Scanning STED FCS), en collaboration avec C. Eggeling (U. Oxford/ Univ Jena, UK-Allemagne) et J Chojnacki (U. Oxford/ IRSI Caixa, Barcelona, SP) (Favard et al., 2019).
Ce travail a été mis en avant par la Company of Biologists, par l‘INSB du CNRS dans ses actualtités et dans ses faits marquants 2019 et par l’ANRS.
2. Microscopie de molécule unique de l’assemblage de HIV-1 dans des lymphocytes T CD4 vivants 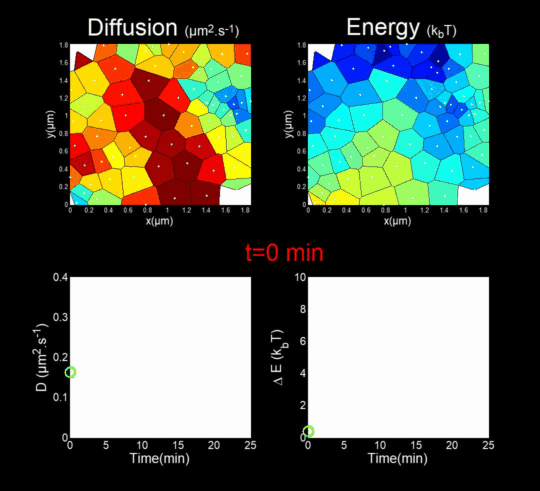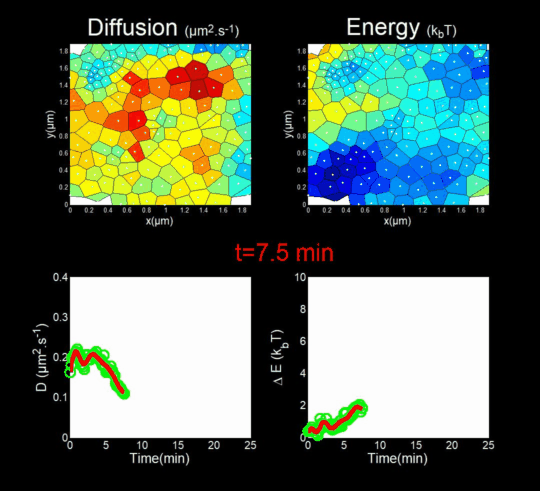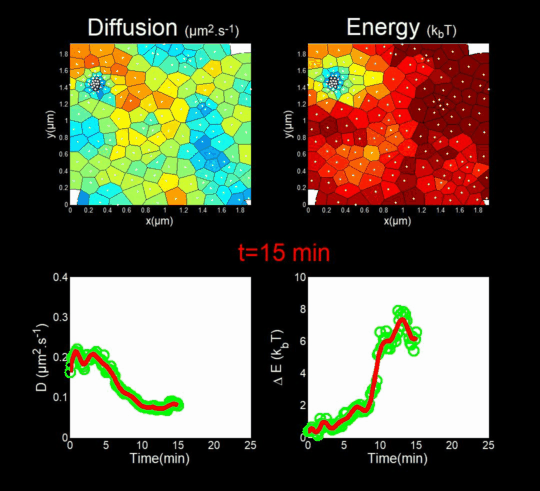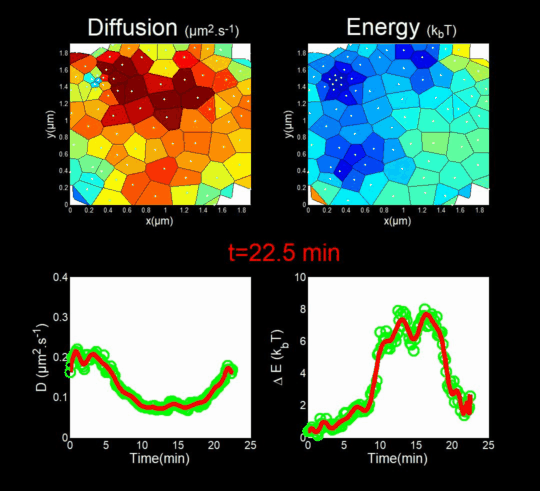
(Financement : ANR Fluobuds, CNRS, Univ Montp, GdR MIV, Erasmus Mundus) En parallèle, nous étudions la dynamique de l’assemblage des protéines de capside dans la cellule par des méthodes de microscopies optiques de super résolution (Live PALM, spt-PALM), en collaboration avec les équipes de JB Sibarita (IINS, Bordeaux) et de M. Dahan (Institut Curie, Paris). Nous retraçons l’assemblage, molécule par molécule, à une résolution de quelques nanométres, générant des milliards de trajectoires individuelles (Mariani et al., 2016, Journal Cover) Nous nous appuyons sur des méthodes issues du Big Data, pour l’analyse automatique de ces trajectoires à l’aide d’inférences Bayésiennes. En collaboration avec J.B. Masson (Institut Pasteur), M. El Beheiry et M. Dahan (I. Curie) nous avons ainsi pu suivre pendant 25 minutes, molécules après molécules l’auto-assemblage de Gag lors de la formation d’une VLP de HIV-1 dans un lymphocyte T. Ceci en mesurant à la fois les changements dans la diffusion de ces molécules (gauche du film) mais aussi dans l’énergie d’attraction (droite du film) qu’elles ressentent à la proximité du site d’assemblage. (Mariani et al., 2018) Nos travaux sur l’étude de l’assemblage du HIV-1 par microscopies de super résolution du vivant ont donné lieu à 2 revues d’intérêt général (Inamdar et al., 2019 et Arone et al., 2021).
3. Dynamique de l’actine corticale et courbure membranaire au cours de l’assemblage viral
Ce projet vise à identifier des co-facteurs cellulaires issus de cellules hôte du HIV-1 (lymphocytes T-CD4+) et capables de moduler de façon concertée, la dynamique, l’organisation de domaines membranaires et la courbure membranaire au cours de l’assemblage du HIV-1 ainsi que l’actine corticale filamenteuse (F-actin). Au cours de nos recherches, nous avons montré que la tétraspanine CD81 est une protéine transmembranaire de la cellule hôte impliquée dans l’assemblage de HIV-1 dans les cellules T CD4 (Grigorov et al., 2009) et que la voie d’endocytose clathrine dynamine était impliquée dans la transmission du HIV-1 des cellules T CD4 aux cellules T CD4 (Bosch et al., 2008). Nos travaux s’intéressent maintenant à l’étude du rôle de ces tétraspanines dans l’assemblage d’un autre rétrovirus humain HTLV-1 infectant chroniquement les lymphocytes T en collaboration avec Hélène Dutartre (CIRI, ENS Lyon) (financement ANRS 2021)
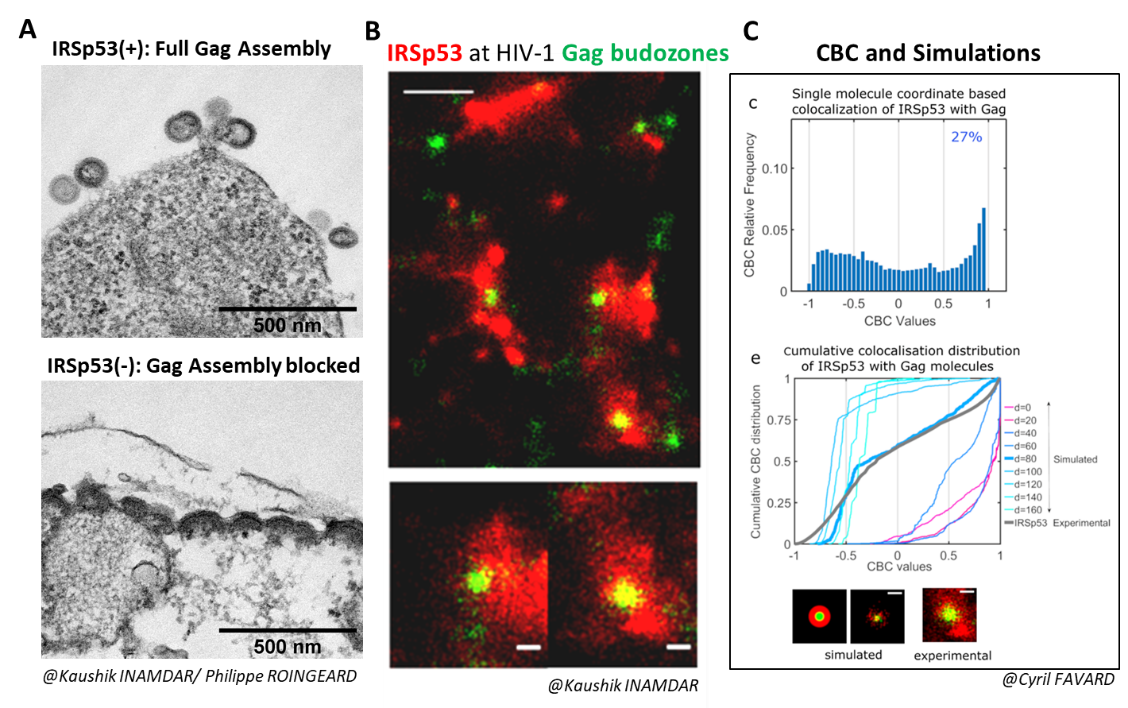
From Inamdar et al., 2021.
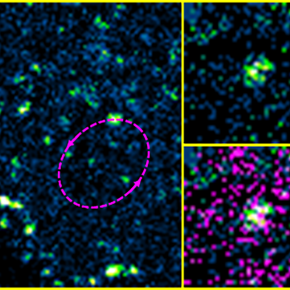
Nous avons aussi étudié le rôle d’un régulateur majeur du réseau d’actine corticale régulé par la voie d’activation Rac1/IRSp53/Wave2/arp2/3 dans l’assemblage de la protéine Gag du HIV-1 et le relargage de la particule virale dans les cellules T CD4, hôtes principales du HIV-1 (Thomas et al., 2015). Nous avons pu montrer qu’effectivement le facteur de courbure membranaire IRSp53 était impliqué dans l’assemblage et le bourgeonnement de la particule virale Gag du HIV-1 (Inamdar et al., 2021). Au cours de la réduction d’expression de IRSp53 par ARN interférent dans des cellules productrice de particules Gag du HIV-1, nous observons un blocage de l’assemblage du virus à mi-parcours (Figure A). IRSp53 est retrouvé au site d’assemblage du virus , comme observé par microscopie PALM (Gag)/STORM (IRSp53 (Figure B) et quantifié en corrélant des mesures expérimentales de single molecule coordinate based colocalisation (CBC) avec celles obtenues par simulations numériques sur des modèles pré-établis (Figure C). La protéine cellulaire IRSp53 aide à la courbure membranaire lors de la formation de la particule de HIV-1 afin de finaliser l’assemblage et le bourgeonnement du HIV-1. D’après Inamdar et al., 2021.
4. Rôle des protéines de matrice M et de cofacteurs cellulaires dans l’assemblage du virus de la grippe et du SARS-CoV-2.
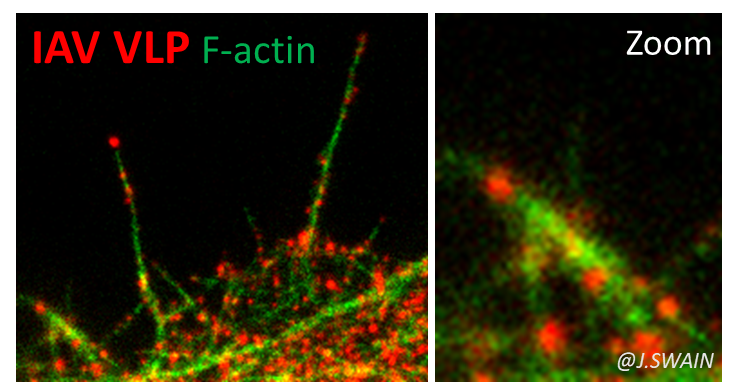 Nous étudions d’autres virus enveloppés à ARN. Le virus de la grippe, A (H1N1) pdm09, toujours circulant, qui a causé la première pandémie du 21e siècle. Nous nous intéressons au rôle de la protéine de matrice M1 (et M2) dans l’assemblage membranaire de ce virus. M1 est la protéine virale la plus abondante et est indispensable pour l’assemblage du virus de la grippe. Nous avons récemment montré que le triplet d’arginines (R76/77/78) localisé sur l’hélice 5 de M1, motif très bien conservé parmi les sous-types de la grippe A, était indispensable à sa localisation à la membrane plasmique des cellules, à l’accrochage membranaire de M1, à son incorporation dans les particules virales et à son infectivité (en collaboration avec O.Moncorgé, IRIM CNRS Montpellier et P. Roingeard, Université de Tours). Sa mutation abolit complètement la production de virus infectieux. Cette étude, nous a également permis d’élaborer un système minimum de production de VLP-M de la grippe A non infectieuses (Kerviel, Dash et al., 2016). Nous nous intéressons maintenant, au rôle de l’actine corticale et de ses régulateurs de sa dynamique dans l’assemblage et le bourgeonnement du virus de la grippe A et du SARS-CoV-2 (Bracquemond & Muriaux, 2021), grâce à des techniques d’ARN interférents, d’infection, et de biologie cellulaire couplée à de la microscopie photonique avancée.
Nous étudions d’autres virus enveloppés à ARN. Le virus de la grippe, A (H1N1) pdm09, toujours circulant, qui a causé la première pandémie du 21e siècle. Nous nous intéressons au rôle de la protéine de matrice M1 (et M2) dans l’assemblage membranaire de ce virus. M1 est la protéine virale la plus abondante et est indispensable pour l’assemblage du virus de la grippe. Nous avons récemment montré que le triplet d’arginines (R76/77/78) localisé sur l’hélice 5 de M1, motif très bien conservé parmi les sous-types de la grippe A, était indispensable à sa localisation à la membrane plasmique des cellules, à l’accrochage membranaire de M1, à son incorporation dans les particules virales et à son infectivité (en collaboration avec O.Moncorgé, IRIM CNRS Montpellier et P. Roingeard, Université de Tours). Sa mutation abolit complètement la production de virus infectieux. Cette étude, nous a également permis d’élaborer un système minimum de production de VLP-M de la grippe A non infectieuses (Kerviel, Dash et al., 2016). Nous nous intéressons maintenant, au rôle de l’actine corticale et de ses régulateurs de sa dynamique dans l’assemblage et le bourgeonnement du virus de la grippe A et du SARS-CoV-2 (Bracquemond & Muriaux, 2021), grâce à des techniques d’ARN interférents, d’infection, et de biologie cellulaire couplée à de la microscopie photonique avancée.
5. Etude par microscopie à force atomique (AFM) de l’assemblage du HIV-1, IAV et SARS-CoV-2 sur des particules virales uniques
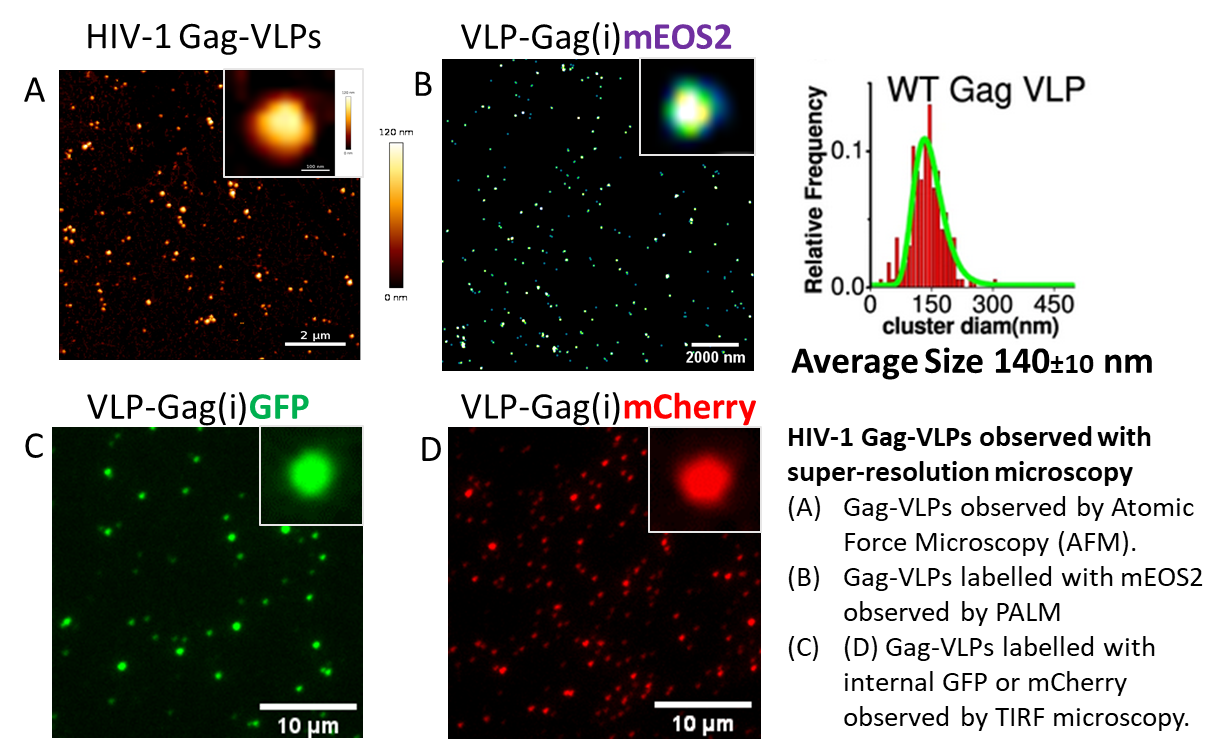 (Financement : ANRS 2015, ANRS 2019, MUSE) Nous avons montré à l’aide de la microscopie à force atomique au niveau de particules virales uniques que l’ARN génomique était un facteur de polydispersité de la morphologie du virus HIV-1 (Faivre-Moskalenko et al.,2014). En collaboration avec les biophysiciens C. Moskalenko et M. Castelnovo à l’ENS de Lyon, la microscopie à force atomique est utilisée pour étudier quantitativement la variation de la taille des particules et des cores viraux produits par des cellules hôtes, en fonction de divers micro-environnements (Bernaud et al., 2015). Cette polydispersité sert d’estimateur de l’efficacité l’auto-assemblage de la protéine Gag de HIV-1. Nous avons récemment imager le virus SARS-CoV2 infectieux et inactivés par AFM en collaboration avec Sébastien lyonnais et l’équipe du Cemipai (Lyonnais et al., 2021). Nous continuons à analyser tous types de VLP dérivés des virus HIV-1, IAV et SARS-CoV-2 par ces techniques de fluorescence, qui représentent de merveilleux outils pour étudier l’assemblage ou l’entrée des virus dans leur cellule hôte (en dehors d’un BSL3).
(Financement : ANRS 2015, ANRS 2019, MUSE) Nous avons montré à l’aide de la microscopie à force atomique au niveau de particules virales uniques que l’ARN génomique était un facteur de polydispersité de la morphologie du virus HIV-1 (Faivre-Moskalenko et al.,2014). En collaboration avec les biophysiciens C. Moskalenko et M. Castelnovo à l’ENS de Lyon, la microscopie à force atomique est utilisée pour étudier quantitativement la variation de la taille des particules et des cores viraux produits par des cellules hôtes, en fonction de divers micro-environnements (Bernaud et al., 2015). Cette polydispersité sert d’estimateur de l’efficacité l’auto-assemblage de la protéine Gag de HIV-1. Nous avons récemment imager le virus SARS-CoV2 infectieux et inactivés par AFM en collaboration avec Sébastien lyonnais et l’équipe du Cemipai (Lyonnais et al., 2021). Nous continuons à analyser tous types de VLP dérivés des virus HIV-1, IAV et SARS-CoV-2 par ces techniques de fluorescence, qui représentent de merveilleux outils pour étudier l’assemblage ou l’entrée des virus dans leur cellule hôte (en dehors d’un BSL3).
6. Etude de la dynamique de réplication virale (traduction/amplification) en cellules et au cours du développement.

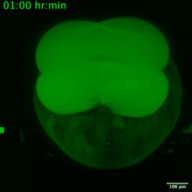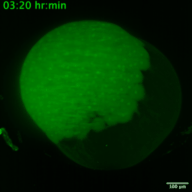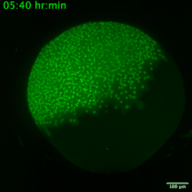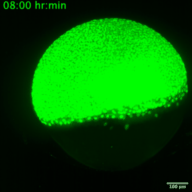 Les virus à ARN représentent un problème majeur de santé publique dans le monde entier. Certains de ces virus à ARN sont impliqués dans des maladies infectieuses émergentes. Alors que l’humanité se remet lentement de la pandémie de COVID-19, l’expansion des virus à vecteur arthropode (arbovirus) nécessite une prise de conscience immédiate pour éviter une nouvelle crise sanitaire mondiale. Les arbovirus appartiennent principalement à deux familles de virus à ARN monocaténaire à sens positif : Flaviviridae et Togaviridae, dont font partie des virus tels que le Zika, le Chikungunya et le Sindbis. Chez l’homme, les symptômes cliniques les plus courants de l’infection sont la fièvre, les maux de tête et les douleurs, mais ils peuvent atteindre le système nerveux et entraîner des encéphalites et des troubles neuronaux. Des recherches fondamentales sur leurs cycles de réplication et la réponse immunitaire associée sont nécessaires et faciliteraient le développement de nouveaux traitements. En effet, la localisation, la traduction et la réplication du génome viral sont des processus complexes et hautement régulés qui se produisent pendant la réplication des virus à ARN. Ces processus sont étroitement orchestrés et leur synchronisation est cruciale pour la formation de nouveaux virus. Bien que les virus à ARN aient été largement étudiés, on sait peu de choses sur les processus spatio-temporels qui ont lieu pendant l’infection virale dans les organismes vivants (par exemple, quelle est la dynamique de la traduction virale ? à quelle échelle de temps les cellules hôtes sont-elles capables de répondre à l’infection ?) De plus, ces dynamiques peuvent varier en fonction du type de cellule et du stade de développement au moment de l’infection. Grâce au développement récent de méthodologies permettant d’étudier en direct la traduction d’ARNm uniques dans un organisme vivant, nous proposons de disséquer, in vivo, l’interaction entre la dynamique de traduction/réplication des virus Zika, Chikungunya et Sindbis et la réponse immunitaire des cellules infectées. Nous utiliserons le poisson zèbre comme organisme modèle pour étudier la dynamique du cycle viral et la réponse immunitaire associée au cours du développement, avec une haute résolution spatio-temporelle et au niveau de la molécule unique.
Les virus à ARN représentent un problème majeur de santé publique dans le monde entier. Certains de ces virus à ARN sont impliqués dans des maladies infectieuses émergentes. Alors que l’humanité se remet lentement de la pandémie de COVID-19, l’expansion des virus à vecteur arthropode (arbovirus) nécessite une prise de conscience immédiate pour éviter une nouvelle crise sanitaire mondiale. Les arbovirus appartiennent principalement à deux familles de virus à ARN monocaténaire à sens positif : Flaviviridae et Togaviridae, dont font partie des virus tels que le Zika, le Chikungunya et le Sindbis. Chez l’homme, les symptômes cliniques les plus courants de l’infection sont la fièvre, les maux de tête et les douleurs, mais ils peuvent atteindre le système nerveux et entraîner des encéphalites et des troubles neuronaux. Des recherches fondamentales sur leurs cycles de réplication et la réponse immunitaire associée sont nécessaires et faciliteraient le développement de nouveaux traitements. En effet, la localisation, la traduction et la réplication du génome viral sont des processus complexes et hautement régulés qui se produisent pendant la réplication des virus à ARN. Ces processus sont étroitement orchestrés et leur synchronisation est cruciale pour la formation de nouveaux virus. Bien que les virus à ARN aient été largement étudiés, on sait peu de choses sur les processus spatio-temporels qui ont lieu pendant l’infection virale dans les organismes vivants (par exemple, quelle est la dynamique de la traduction virale ? à quelle échelle de temps les cellules hôtes sont-elles capables de répondre à l’infection ?) De plus, ces dynamiques peuvent varier en fonction du type de cellule et du stade de développement au moment de l’infection. Grâce au développement récent de méthodologies permettant d’étudier en direct la traduction d’ARNm uniques dans un organisme vivant, nous proposons de disséquer, in vivo, l’interaction entre la dynamique de traduction/réplication des virus Zika, Chikungunya et Sindbis et la réponse immunitaire des cellules infectées. Nous utiliserons le poisson zèbre comme organisme modèle pour étudier la dynamique du cycle viral et la réponse immunitaire associée au cours du développement, avec une haute résolution spatio-temporelle et au niveau de la molécule unique.
Responsables

Delphine Muriaux
Directrice CEMIPAI,
DR1 CNRS, HDR
Virologie

Cyril Favard
IRHC CNRS, HDR
Biophysique

Nous étudions les mécanismes de l’assemblage des virus enveloppés, de la molécule unique aux organoïdes.
Notre équipe est pluridisciplinaire et fait appel à la biologie cellulaire, la biochimie, la biophysique et la biophotonique.
Financements





























 Rayane a brillament été sélectionnée pour présenter ses travaux sur le rôle de l’actine durant l’assemblage de HIV-1. De plus elle a obtenue une bourse de SIDACTION pour financer son déplacement au congrès.
Rayane a brillament été sélectionnée pour présenter ses travaux sur le rôle de l’actine durant l’assemblage de HIV-1. De plus elle a obtenue une bourse de SIDACTION pour financer son déplacement au congrès.