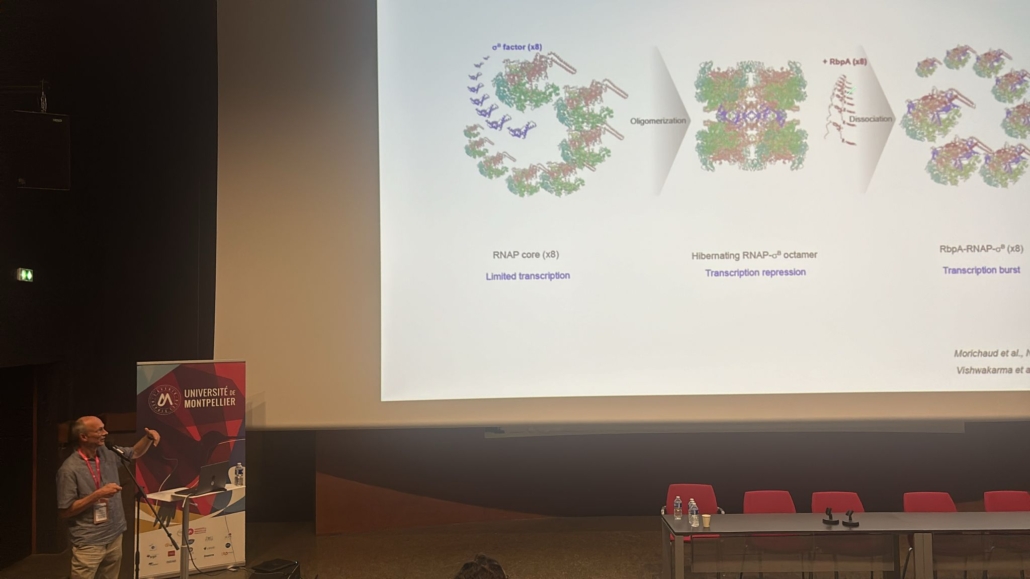
Axe 1: Bacterial RNA polymerase and antibiotic resistance (K. Brodolin)
L’ADN-dépendante polymerase de l’ARN (ARNP) est l’enzyme centrale de l’expression génique et une cible pour la régulation génétique. Ses caractéristiques de base sont hautement conservées parmi tous les organismes vivants. l’ARNP bactérienne est une cible pour un grand nombre de protéines de régulation et de petites molécules, notamment des antibiotiques. Parmi les molécules antibactériennes inhibant l’ARNP, la rifampicine (Rif) reste un antibiotique de première ligne pour le traitement de la tuberculose. Récemment, une nouvelle classe d’antibiotiques: lipiarmycin (fidaxomicine, Tiacumicin B) et myxopyronin (Myx) a été caractérisée. Le mécanisme d’action de ces molécules reste à élucider. Les bactéries ont développé des mécanismes multiples pour échapper aux effets du traitement antibiotique. La plupart de ces mécanismes, tels que l’activation des voies de réponse au stress et le passage à l’état persistant sont liées à la régulation de l’activité de l’ARNP. Une quantité croissante de preuves indique que la sous-unité sigma de l’ARNP ainsi que d’autres facteurs transcriptionnels (par exemple la RbpA de M. tuberculosis) sont impliqués dans la génération de la résistance en réponse au traitement antibiotique. Les objectifs globaux de nos études sont (1) comprendre la rôle de l’interaction entre la sous-unité sigma et les facteurs transcriptionnels dans la modulation de l’expression génique; (2) déchiffrer les mécanismes d’action des antibiotiques ciblant l’ARNP (3) comprendre comment les bactéries résistent à ces molécules. Notre projet est axé sur le mécanisme de régulation de la transcription de deux agents pathogènes humains: Escherichia coli, qui est considéré comme un prototype pour les bactéries pathogènes, et Mycobacterium tuberculosis qui est d’une grande importance clinique. Nous explorons également le rôle de la sous-unité sigma dans les étapes clés de l’expression des gènes tels que la reconnaissance de promoteur, la synthèse de l’ARN et pause de la transcription. Les résultats de notre étude contribuent à la compréhension de la base moléculaire de la résistance aux antibiotiques, constituent une base pour le développement de nouveaux médicaments, plus efficaces en pharmacologie.
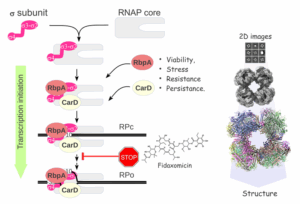
Axe 2: Development of new anti-infectious therapeutic strategies (L. Chaloin)
The aim of our studies is to develop new therapeutic weapons to combat certain viral and bacterial infections more effectively. A related theme is to gain a better understanding of the action of antibiotics directed against RNA polymerase, in particular the repositioning of Fidaxomicin (initially developed against Clostridium difficile) to inhibit transcription in M. tuberculosis. Biochemical, structural and theoretical studies are carried out in parallel to elucidate the mechanism of action, the emergence of resistance and the development of new antibiotics.
Two other themes are being developed in collaboration with the APIR team (J.-M. Péloponèse) to fight against RNA viruses (HIV-1, HTLV-1, Chikungunya, Dengue and SARS-CoV-2) by targeting RNA helicase A (RHA or DHX9), which is used and necessary for the multiplication of these viruses. This project has been now extended to DNA viruses such as hepatitis B virus (HBV) and the study of the role of RHA in the replication of HBV. This work is mainly accomplished by L. Marty during her PhD.
In parallel, a second approach is aimed at halting the transformation of cells infected with HTLV-1, leading to adult T-cell leukemia. This strategy involves developing specific inhibitors of the viral protein HBZ (HTLV-1 bZip factor), which is encoded by the antisense strand of the viral genome and is expressed throughout the infection process, as well as after leukemic transformation, making it a prime target.
For these applied research projects, methodologies such as virtual screening and molecular dynamics simulations are used alongside experimental data obtained within the team or in collaboration with IRIM’ teams.
POST-DOC, THÈSES, STAGES
Nous accueillons des candidats (biologistes, biochimistes, biophysiciens, physico-chimistes, physiciens) mais aussi des chercheurs et des ingénieurs et techniciens en poste (CNRS, INSERM, Université) intéressés par des approches interdisciplinaires de problèmes biologiques à l’échelle de la molécule unique ou d’assemblages moléculaires..
At a glance
Organism(s)
Escherichia coli, Mycobacterium tuberculosis
Biological processes studied
Transcription, antibiotic resistance
Methodologies
- Biochemistry
- Biophysics (x-ray crystallography, electron microscopy)
- Bacteriology
- Molecular modeling and drug design
Medical applications
- Developement of new anti-infectious compounds
Financements